Moral (2023) desarrolló la prueba Q de normalidad multivariante que presenta unas propiedades de potencia y eficiencia adecuadas. En su formulación original, se planteó un cálculo aproximado del valor de probabilidad y potencia usando la distribución chi-cuadrado. No obstante, se sugería definir un enfoque bootstrap más flexible en supuestos.
La prueba Q se formuló con dos variantes; una se calcula con la estandarización de Royston (1992) de las estadísticas de normalidad univariante de Shapiro y Wilk (1965) y otra con la estandarización de Royston (1993) de las estadísticas de normalidad univariante de Shapiro y Francia (1972). Se denotan respectivamente QSWa y QSFa. En el estudio original de simulación, se halló una ligera ventaja de QSWa sobre QSFa, cuyo rendimiento se asemejó a la prueba H de Royston (1983).
La variante QSW se basa en los estadísticos W estandarizados, cuyo cálculo resulta más complejo que el de los estadísticos W’ estandarizados utilizados en la variante QSF. Además, la variante QSF está limitada a muestras de entre 3 y 2000 datos, mientras que la variante QSW puede aplicarse a muestras que van de 5 a 5000 datos. No obstante, la versión QSW presenta una ligera ventaja sobre la QSF en contextos con muestras pequeñas (20 participantes) y pocas variables (4) desde la aproximación chi-cuadrado o versión original de la prueba Q (Moral, 2023). El estudio del enfoque bootstrap para la prueba Q está en proceso.
El objetivo del presente artículo metodológico es desarrollar un script en español para el programa R (R Core Team, 2025a) que permita aplicar la prueba Q. El script abarca tanto la aproximación chi-cuadrado de la propuesta original (QSWa y QSFa) como el enfoque bootstrap sugerido en la misma (QSWb y QSFb). Aparte, se incluye la comprobación de la independencia serial de la secuencia de las estadísticas de normalidad univariante estandarizadas de las 2k-1 posibles combinaciones lineales no ponderadas de las k variables originales (asunción del enfoque asintótico) y la prueba de normalidad multivariante H de Royston (1983). A su vez, el script contiene el cálculo de la potencia tanto desde la aproximación chi-cuadrado como desde el enfoque bootstrap.
La ejecución del script se ilustra con una muestra simulada de 36 observaciones, creada con el generador de números aleatorios de Excel. Los datos simulados corresponden a las puntuaciones de seis variables, cada una con un rango discreto de 1 a 10, de 36 estudiantes de secundaria (18 mujeres y 18 hombres). Estas variables están altamente correlacionadas, ya que conforman una escala unidimensional de rendimiento atlético evaluado por terceros (entrenadores).
La suma de las seis variables define la puntuación total de ejecución atlética, que en otras poblaciones similares se ha demostrado que sigue una distribución normal. Se espera que la hipótesis nula de normalidad multivariante se sostenga con las cuatro variantes de la prueba Q de Moral (2023) y la prueba H de Royston (1983).
Se escogió el programa R por ser uno de los programas más completos de estadística, de acceso abierto y en constante desarrollo y revisión por la comunidad matemática (Braun, & Murdoch, 2021; Giorgi et al., 2022). Se incluyó la prueba H de Royston (1983) por ser una de las pruebas de normalidad multivariante más usada y potente (Anis et al., 2021; Khatun, 2021). Dado que en el estudio original (Moral, 2023) se observó que la proporción de aciertos y la potencia media de la prueba Q eran superiores a las de la prueba K² de Mardia (1980), y se aproximaban a las de la prueba H de Royston (1983), la prueba de Mardia no fue incorporada en los scripts.
Desarrollo del script para la prueba Q de normalidad multivariante
Para obtener la estadística de contraste de la prueba de normalidad multivariante propuesta por Moral (2023), se calculan las estadísticas estandarizadas de la prueba de normalidad univariante de Shapiro y Wilk (1965) (Ecuación 1) o de la prueba de Shapiro y Francia (1972) (Ecuación 2), utilizando la estandarización desarrollada por Royston (1992, 1993), de todas las posibles combinaciones lineales no ponderadas (nc) entre las k variables, lo que equivale a un número de 2k – 1 combinaciones (Ecuación 3). En la primera variante, la estadística de contraste se denota por QSW como variable aleatoria y q como valor específico. En la segunda variante, se simboliza por QSF como variable aleatoria y q‘ como valor específico.
Los valores negativos de z o z’ se truncan a 0 para evitar una sobreestimación de QSW o QSF, ya que un valor negativo de z o z’ implica un buen ajuste a la normalidad, al calcularse el valor de probabilidad a la cola derecha en una distribución normal estándar. Véanse las ecuaciones 4 y 5.
Una vez que se tienen las 2k – 1 estadísticas estandarizadas y se han truncado a 0 los negativos, se calcula su suma de cuadrados y se obtiene el valor de probabilidad a la cola derecha en una distribución chi-cuadrado. En la propuesta original (Moral, 2023), los grados de libertad (gl) son el número de valores sumados (nc) menos el número de valores truncados (a): gl = nc – a (propuesta original). Véase Ecuación 6.
En el script de este estudio, restar los valores truncados se deja como opción. En el estudio de simulación no se usa, de ahí que la opción por defecto es que los grados de libertad sea el número de combinaciones (nc). Debido a que la verdadera distribución muestral de la estadística de contraste corresponde a una distribución chi-cuadrado generalizada, la cual es menos apuntada y presenta una asimetría positiva mayor que la distribución chi-cuadrado estándar, se consideró una opción más adecuada (Rolke & Gongora, 2021).
Dado que la aproximación asintótica basada en la distribución chi-cuadrado estándar para la estadística de contraste asume no solo la normalidad de las variables sumadas, sino también su independencia (Chen & Lumley, 2019), se evalúa la independencia serial de dichos valores mediante la prueba de rachas de Wald y Wolfowitz (1943), utilizando el paquete DescTools de R (Signorell et al., 2025), y la prueba de Ljung y Box (1978), implementada en el paquete stats de R (R Core Team, 2025b). Esta última se añade debido a que permite especificar el rezago máximo (h) con el que se desea realizar el análisis (de 1 a h), que en el caso de la prueba de Wald y Wolfowitz (1943) es de 1.
En series no estacionales o sin un patrón temporal esperado, se recomienda analizar rezagos desde 1 hasta el menor de 10 o un quinto del número de elementos en la serie (Chan et al., 2022; Hyndman & Athanasopoulos, 2021). También se incluye como opción la regla de Schwert (1989) para la determinación del rezago máximo, que es ampliamente usada (Schmal, 2024). Véase ecuación 7.
Adicionalmente, el script contiene la aproximación bootstrap como alternativa para el cálculo del valor de probabilidad y la potencia estadística (Egbert & Plonsky, 2021; Rousselet et al., 2023), especialmente útil si se incumple el supuesto de independencia en la secuencia de las 2k-1 combinaciones lineales. Por una parte, se obtiene la distribución muestral de la estadística de contraste Q o Q’ a partir de 1000 muestras extraídas por muestreo con reemplazamiento (bootstrap) desde la muestra original de n observaciones para k variables correlacionadas (distribución bootstrap empírica de la estadística Q o Q’).
Por otra parte, se genera la distribución muestral normativa de la estadística de contraste Q o Q’ a partir de 1000 muestras obtenidas mediante muestreo con reposición (bootstrap), extraídas de una muestra de tamaño n compuesta por k variables normalmente distribuidas y que conservan la misma estructura correlacional que la muestra original, denominada muestra normativa. De este modo, se aplica, por una parte, un procedimiento bootstrap no paramétrico y, por otra, uno paramétrico bajo la hipótesis nula de normalidad multivariante.
Para generar la población o muestra fuente en el bootstrap paramétrico, se emplea la matriz diagonal inferior de la factorización de Cholesky de la matriz de correlaciones de las variables originales como un factor de postmultiplicación. Una matriz (generada) de tamaño n × k, compuesta por vectores gaussianos independientes, actúa como el factor de premultiplicación en este producto matricial.
El resultado es una matriz compuesta por k vectores gaussianos n-dimensionales correlacionados (Marchev & Marchev, 2024; Ripley et al., 2025). Las puntuaciones de los k vectores gaussianos (de n puntuaciones con distribución normal estándar) se generan a partir de órdenes de cuantil equiespaciados, que van de .5/n a 1 – .5/n, aplicados a la función probit, es decir, a la función cuantil de la distribución normal estándar. Los órdenes se permutan, usando una semilla (123) para que el resultado sea estable. De este modo, cada una de las k variables se crea con los mismos órdenes de cuantil, pero en una secuencia diferente, por lo que resultan vectores independientes.
Al extraer aleatoriamente, con reemplazamiento, 1000 muestras de la muestra fuente y calcular la estadística de contraste en cada una de ellas, se obtiene la denominada distribución normativa, la cual permite computar la probabilidad bootstrap y obtener el valor crítico bootstrap (cuantil de orden .95) para el cálculo de la potencia estadística.
El valor de probabilidad bootstrap (proporción de valores mayores o iguales que el valor de la estadística de contraste q o q’) se calcula a partir de la distribución bootstrap normativa (chi-cuadrado generalizada) como sustitución de la distribución chi-cuadrado estándar.
La potencia estadística (ϕ) para un nivel de significancia α se obtiene como la probabilidad (proporción) de rechazos en la muestra bootstrap empírica, determinando la región de rechazo con el cuantil 1-α de la distribución bootstrap normativa. En ambos casos, el remuestreo con reposición se realiza por filas, en lugar de por columnas, con el fin de preservar las correlaciones entre las variables de la muestra fuente. Para que los resultados sean estables, se usa una semilla (123) cuando se generan las 1000 muestras bootstrap.
Por otra parte, se determina la mediana de la distribución bootstrap normativa para calcular la probabilidad asociada a este valor central en la cola izquierda y cola derecha de la distribución de la muestra bootstrap empírica. El doble del menor de estos dos valores representa la probabilidad de que la distribución bootstrap empírica esté centrada en la mediana de la distribución bootstrap normativa. Cuanto más próximo a 1 sea dicho valor de probabilidad, mayor será la evidencia de equivalencia de medianas entre las distribuciones bootstrap teórica y empírica; en cambio, un valor menor que .05 indicará un claro descentramiento de la distribución bootstrap empírica con respecto a la normativa.
El alejamiento de la distribución bootstrap empírica de la normativa revela desviación de la normalidad multivariante. Cabe señalar que los valores q y q’ normativos debe ser valores más próximos a 0 que al número de combinaciones de las k variables originales y alejados de la mediana de la distribución bootstrap normativa.
Adicionalmente, se representan ambas distribuciones mediante un histograma y curvas de densidad, con el fin de evaluar si la distribución muestral de la estadística se ajusta efectivamente a una distribución chi-cuadrado estándar (curva de referencia) o si adopta una forma chi-cuadrado generalizada, característica de formas cuadráticas o sumas de cuadrados de variables correlacionadas (Goetze & Tikhomirov, 2002). Dicha distribución tiene un pico más bajo y una cola derecha más pesada que la distribución chi-cuadrado estándar. El número de intervalos de clase (k) y su amplitud constante (a) se calcula mediante la regla de la Universidad Rice (Ecuación 8) que es adecuada para muy diversas distribuciones, incluidas aquellas con asimetría y leptocurtosis (Moral, 2024).
donde qi o q′i (i = 1, 2, …, 1000) representan las estimaciones del estadístico de contraste en las 1000 muestras bootstrap extraídas de la muestra original de tamaño n.
En el Apéndice 1, se encuentra el script para las dos variantes (aproximación chi-cuadrado y bootstrap) de la prueba Q de normalidad multivariante calculada con las estadísticas de normalidad univariante de Shapiro y Wilk (1965) estandarizadas por el método de Royston (1992) para muestras de tamaño de 12 a 2000 con k mediciones (QSW).
En el Apéndice 2, se halla el script para las dos variantes (aproximación chi-cuadrado y bootstrap) de la prueba Q calculada con las estadísticas de normalidad univariante de Shapiro y Francia (1972) estandarizadas por el método de Royston (1993) para muestras de tamaño de 5 a 5000 con k de mediciones (QSF).
Las dos variantes de la prueba QSW o QSF de Moral (2023) se complementan con la prueba de normalidad multivariante H de Royston (1983), que se encuentra en el Apéndice 3, para muestras de tamaño de 10 a 2000 con k mediciones. Se usa un nivel de significancia (α) de .05 para la prueba H y la aproximación chi-cuadrado de la prueba Q y .1 para el enfoque bootstrap de la prueba Q, lo que se destaca en verde.
Finalmente, se incluye el cálculo de la potencia relativa entre la prueba Q (evaluada mediante la aproximación chi-cuadrado o el enfoque bootstrap) y la prueba H (Royston, 1983). Para implementar correctamente este último análisis del Apéndice 3, es necesario ejecutar previamente el script del Apéndice 1 (estadísticas W de Shapiro y Wilk, 1965, estandarizadas por Royston, 1992) o el del Apéndice 2 (estadísticas W’ de Shapiro y Francia, 1972, estandarizadas por Royston, 1993). Dichos scripts generan los valores de power (potencia bajo la aproximación chi-cuadrado) y boot_power (potencia obtenida mediante bootstrap). Después de ello, debe correrse el script del Apéndice 3, que calcula power_H para la prueba H. Alternativamente, es posible introducir manualmente los valores de potencia estadística obtenidos en una ejecución previa e independiente de los Apéndices 1 o 2.
Para ejecutar los scripts es necesario tener instalados y cargados los paquetes nortest (Gross &, Ligges, 2022), MASS (Ripley, et al., 2025), DescTools (Signorell, 2025), stats (R Core Team, 2025b) y MVN (Korkmaz, 2025) del programa R (R Core Team, 2025a). Se recomienda utilizar la última versión de R. Cada script puede ser ejecutado de forma independiente, ya que cuenta con su lista de datos y la llamada de los paquetes requeridos.
Aplicación del script de las pruebas Q de Moral y H de Royston
El script se aplicó a seis variables con 36 observaciones (muestra simulada); no obstante, puede adaptarse a otros conjuntos de datos modificando la lista de variables, que se destaca en color azul. Puede ejecutarse tanto en una computadora con R instalado como en línea en el sitio snippets, disponible en la dirección: https://rdrr.io/snippets/ Si se quiere guardar el gráfico como un archivo JPG, se deben quitar los símbolos del numeral (hashtag) de las instrucciones: # jpg(), # par() y # dev.off(). Véase Apéndices 1‑3. En la Tabla 1, se resumen las condiciones de simulación.
Tabla 1
Condiciones de simulación
Prueba QSW desde la aproximación chi-cuadrado y el enfoque bootstrap
Tras ejecutar el script del Apéndice 1, se obtienen los siguientes resultados:
Tamaño muestral: n = 36.
Número de variables en la muestra original: k = 6.
Media de las correlaciones entre las variables: m(R) = .7127.
Nivel de significancia: α = .05 en la aproximación chi-cuadrado y .1 en el enfoque bootstrap para compensar la cola muy larga a la derecha.
Parámetros comunes para la estandarización de las 63 variables creadas por combinación lineal de las seis variables originales (Tabla 1):
Valor esperado de ln(1-W): μ = -3.596347.
μ = 0.0038915 × ln³(n) – 0.083751 × ln²( n) – 0.31082 × ln(n) – 1.5861
= 0.0038915 × ln³(20) – 0.083751 × ln²(20) – 0.31082 × ln(20) – 1.5861 = -3.164227.
Desviación estándar de ln(1-W): σ = 0.4782324.
σ = e(0.0030302 × ln²(n) – 0.082676 × ln(n) – 0.4803) = e(0.0030302 × ln²(20) – 0.082676 × ln(20) – 0.4803) = 0.4961977.
Número de combinaciones entre las seis variables originales: nc = 63.
Lista de las 63 combinaciones lineales entre las seis variables (Tabla 1):
c1 = x1, c2 = x2, c3 = x3, c4 = x4, c5 = x5, c6 = x6,
c7 = x1 + x2, c8 = x1 + x3, c9 = x1 + x4, c10 = x1 + x5, c11 = x1 + x6,
c12 = x2 + x3, c13 = x2 + x4, c14 = x2 + x5, c15 = x2 + x6,
c16 = x3 + x4, c17 = x3 + x5, c18 = x3 + x6, c19 = x4 + x5, c20 = x4 + x6, c21 = x5 + x6,
c22 = x1 + x2 + x3, c23 = x1 + x2 + x4, c24 = x1 + x2 + x5, c25 = x1 + x2 + x6,
c26 = x1 + x3 + x4, c27 = x1 + x3 + x5, c28 = x1 + x3 + x6, c29 = x1 + x4 + x5,
c30 = x1 + x4 + x6, c31 = x1 + x5 + x6, c32 = x2 + x3 + x4, c33 = x2 + x3 + x5,
c34 = x2 + x3 + x6, c35 = x2 + x4 + x5, c36 = x2 + x4 + x6, c37 = x2 + x5 + x6,
c38 = x3 + x4 + x5, c39 = x3 + x4 + x6, c40 = x3 + x5 + x6, c41 = x4 + x5 + x6,
c42 = x1 + x2 + x3 + x4, c43 = x1 + x2 + x3 + x5, c44 = x1 + x2 + x3 + x6,
c45 = x1 + x2 + x4 + x5, c46 = x1 + x2 + x4 + x6, c47 = x1 + x2 + x5 + x6,
c48 = x1 + x3 + x4 + x5, c49 = x1 + x3 + x4 + x6, c50 = x1 + x3 + x5 + x6,
c51 = x1 + x4 + x5 + x6, c52 = x2 + x3 + x4 + x5, c53 = x2 + x3 + x4 + x6,
c54 = x2 + x3 + x5 + x6, c55 = x2 + x4 + x5 + x6, c56 = x3 + x4 + x5 + x6,
c57 = x1 + x2 + x3 + x4 + x5, c58 = x1 + x2 + x3 + x4 + x6, c59 = x1 + x2 + x3 + x5 + x6,
c60 = x1 + x2 + x4 + x5 + x6, c61 = x1 + x3 + x4 + x5 + x6, c62 = x2 + x3 + x4 + x5 + x6,
c63 = x1 + x2 + x3 + x4 + x5 + x6.
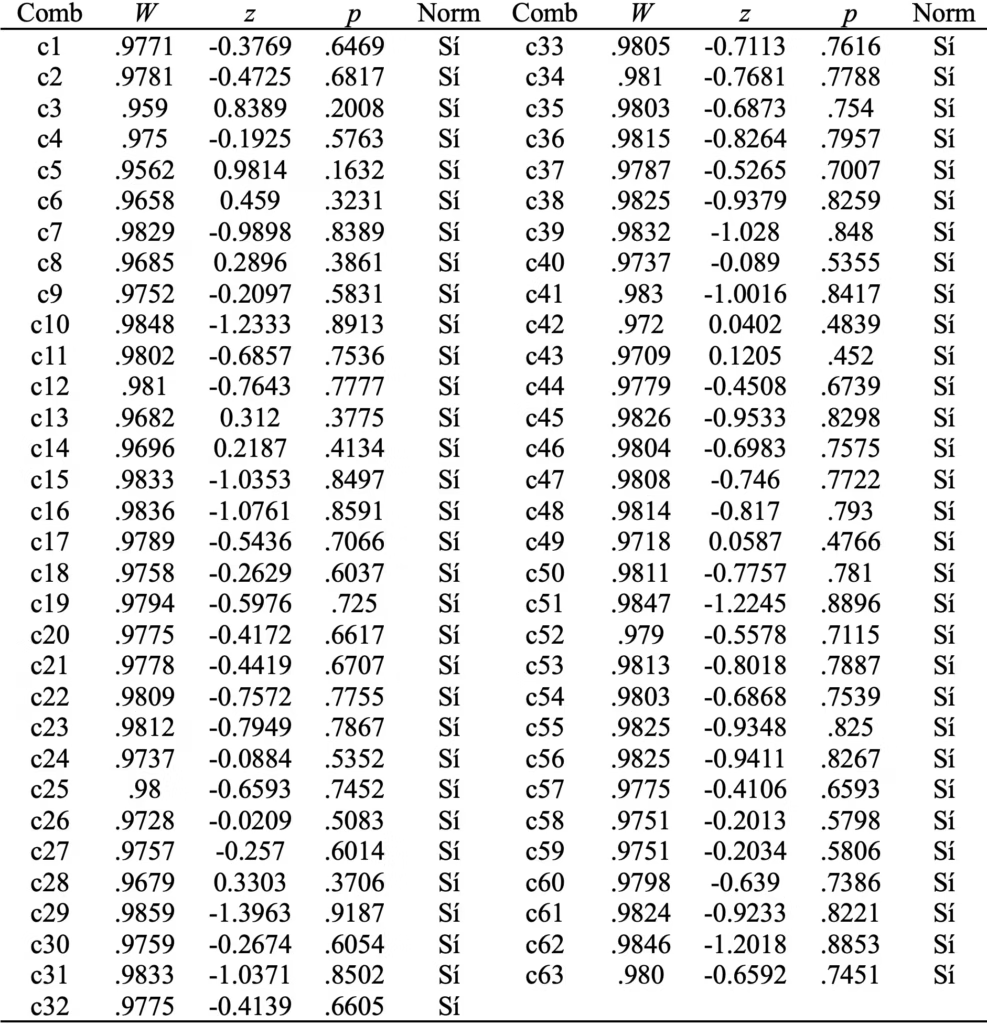
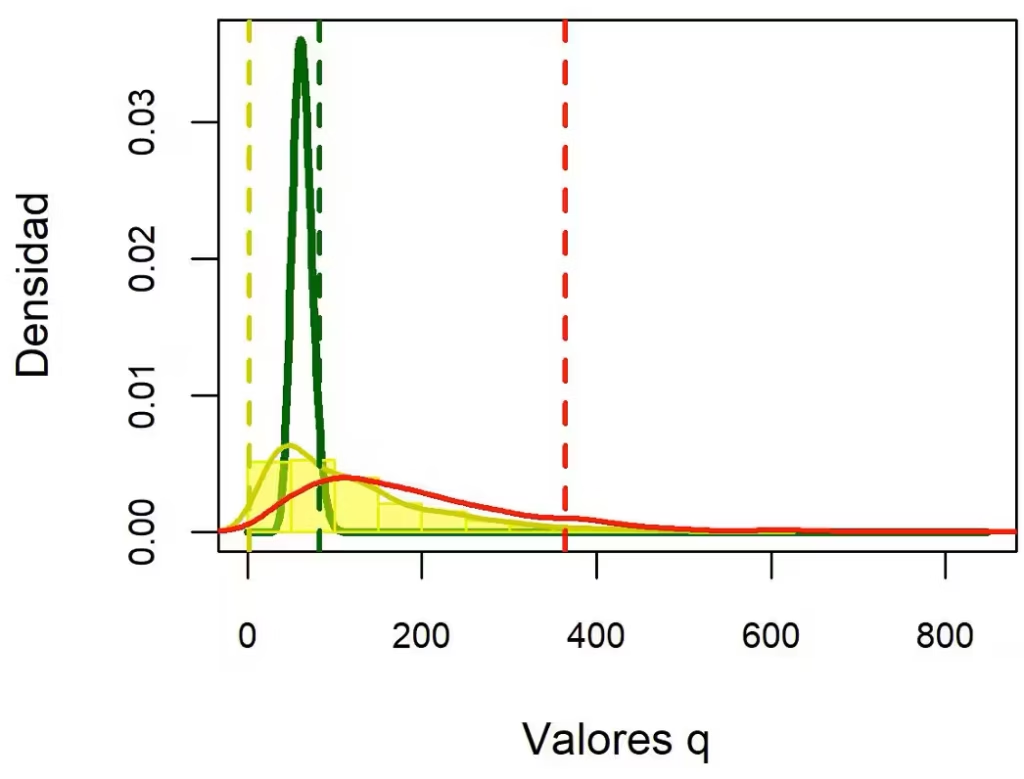
En la Tabla 2, se puede observar que la hipótesis nula de normalidad univariante se sostiene en las 63 combinaciones lineales por la prueba de Shapiro y Wilk (Royston, 1992) con un nivel de significancia del 5%.
Tabla 2
Prueba de normalidad univariante de Shapiro y Wilk con la estandarización de Royston para las 63 combinaciones lineales de las seis variables.
Número de valores z anulados por ser negativos: a = 53.
Grados de libertad: gl = nc = 63. No se restan los valores fijados a 0 (a) para compensar la cola derecha muy larga de la distribución muestral, que permite determinar el enfoque bootstrap. Si se opta por restar a, los grados de libertad son 10.
Estadística de contraste: Q_SW = ∑z² = 2.2353.
Valor de probabilidad y valor crítico desde la aproximación distribución chi-cuadrado estándar:
Valor crítico o cuantil de orden .95 de una distribución chi-cuadrado estándar con 63 grados de libertad: .95χ²[63] = 82.5287. Dado que q = 2.2353 < .95χ²[93] = 82.5287, se mantiene la hipótesis nula de normalidad multivariante a un nivel de significancia de .05 con base en el valor crítico asintótico.
Valor de probabilidad a la cola derecha en una distribución chi-cuadrado estándar con 63 grados de libertad: p = 1 > α = .05. Consecuentemente, la hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de .05 con base en el valor de probabilidad asintótico.
Potencia estadística desde la aproximación chi-2 para un nivel de significancia de .05: phi = .0760 (< .2).
Si no restara el número de valores anulados, igualmente se mantendría la hipótesis nula (q = 2.2353 < .95χ²[10] = 18.307, p = .9942 > .05) con una potencia estadística muy baja (ϕ = .1308 < .2).
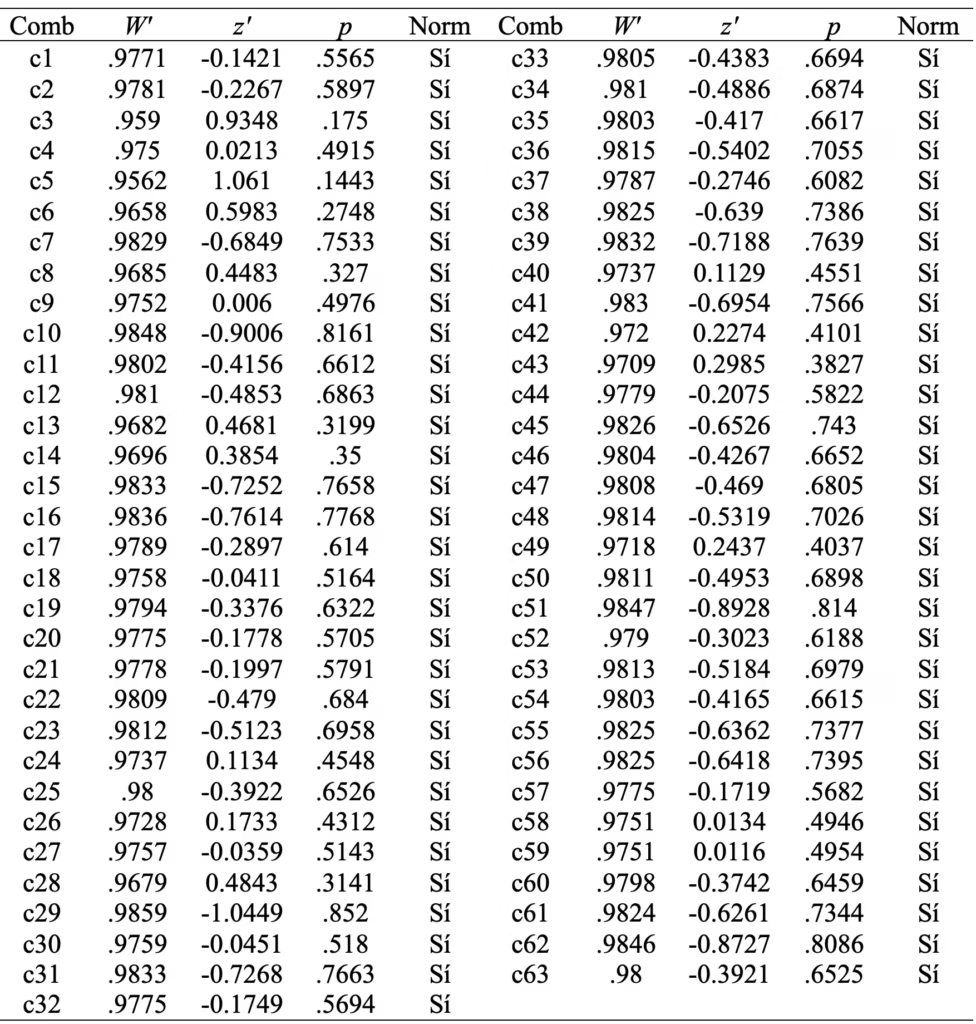
En la secuencia de 63 valores estandarizados de W y truncados a 0 cuando son negativos (a = 53), usados para calcular la estadística de contraste q (z_w), no hay dependencia serial con base en la prueba de rachas de Wald y Wolfowitz (1943) y en la prueba de Ljung y Box (1978) con regazo de 1. No obstante, esta última prueba sí revela dependencia serial con rezagos de 2 a 10 (Tabla 3). El rezago máximo se establece por una regla general para series no estacionarias: min(10, nc/5) = min(10, 63/5) = 10 (Chan et al., 2022). Por la regla de Schwert (1989), el rezago máximo sería 11.
Contraste de la independencia entre los 63 valores de W logarítmicamente transformados, estandarizados y truncados (a 0 cuando son negativos), usados para calcular q(z_w).
Runs test for randomness: data: z_w: runs = 15, m = 53, n = 10, p = .2747 > α = .05.
Tabla 3
Prueba de Ljung y Box de independencia serial en la secuencia de valores z_w
Distribución bootstrap (con correlación conservada) empírica o desde la muestral original:
Número de muestras bootstrap: B = 1000.
Valor de probabilidad bootstrap a la cola derecha en la distribución bootstrap empírica: pboot = .865 > α = .1. Consecuentemente, la hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de .1 con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap empírica.
Distribución bootstrap normativa (con correlación conservada):
Número de muestras bootstrap: B = 1000.
Valor de la estadística q en la distribución bootstrap normativa: qmvn_boot = 11.4279.
Probabilidad de que la distribución bootstrap empírica esté centrada en la mediana de la distribución bootstrap normativa: p2 colas = .496 > α = .1. Consecuentemente, se mantiene la hipótesis nula de medianas equivalentes entre las distribuciones bootstrap teórica y empírica en un contraste a dos colas con un nivel de significancia de .1.
Valor crítico bootstrap o cuantil de orden .9 de la distribución bootstrap normativa: qmvn_crit = 189.5297. Dado que q = 11.42791 < qmvn_crit = 189.5297, se mantiene la hipótesis nula de normalidad multivariante a un nivel de significancia de .1 con base en el valor crítico bootstrap (normativo).
Valor de probabilidad a la cola derecha en la distribución bootstrap normativa: pmvn_boot = 1 > α = .1. Consecuentemente, la hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de .1 con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap normativa.
Potencia estadística bootstrap para un nivel de significancia de .1: phi_boot = .027.
Con un nivel de significancia de .05, la conclusión sería la misma. El valor crítico bootstrap o cuantil de orden .95 de la distribución bootstrap normativa (q_crit) es 418.1868. Así, q = 11.42791 < qmvn_crit = 418.1868. El valor de probabilidad a la cola derecha en la distribución bootstrap normativa (p_mvn_boot) es 1 > α = .05. La potencia estadística bootstrap para un nivel de significancia de .05 (phi_boot) es muy baja (ϕ = .014 < .2).
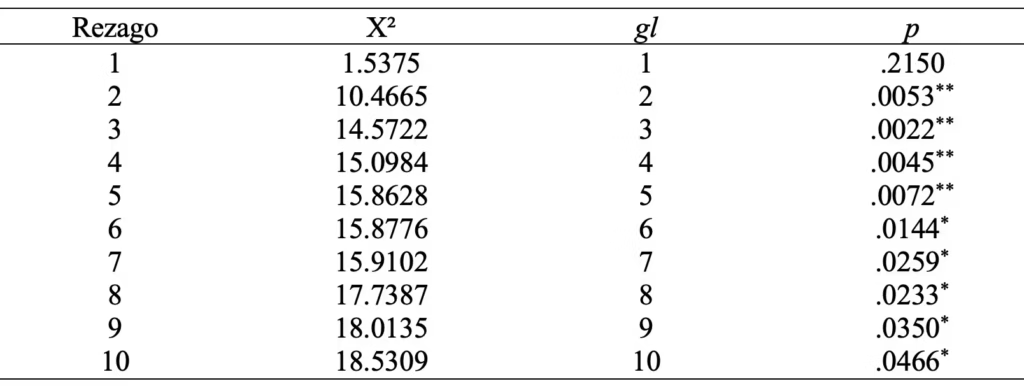
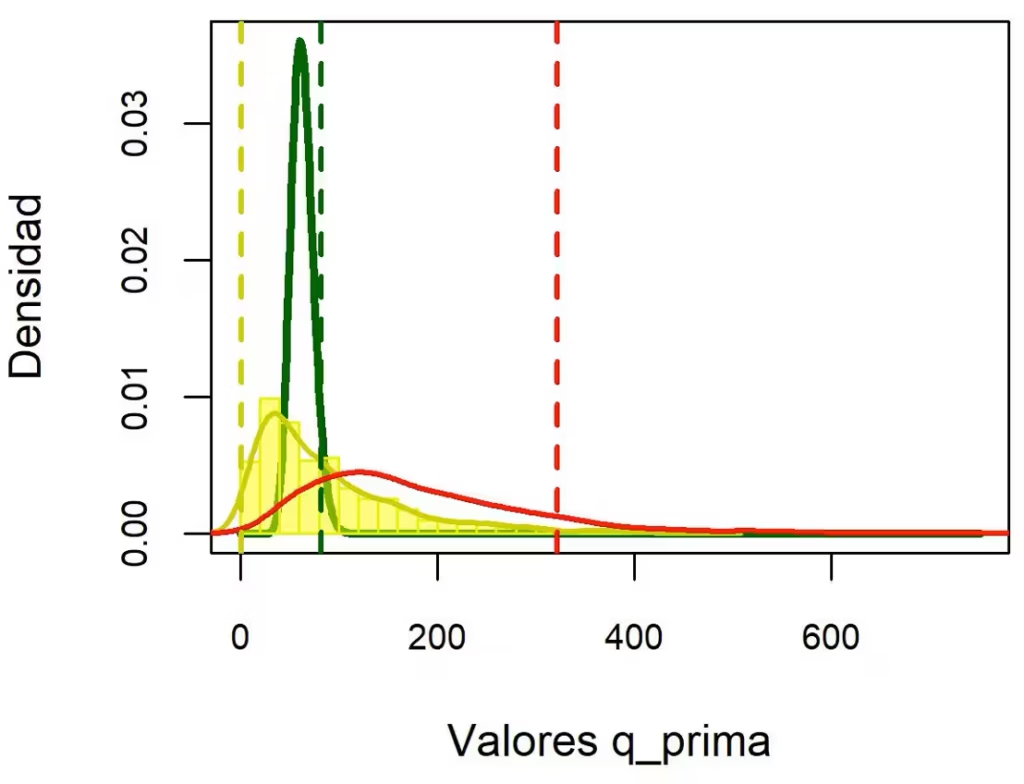
En la Figura 1, el histograma amarillo representa las densidades de la distribución bootstrap empírica de la estadística de contraste q. El número de intervalos de clase se determinó mediante la regla de la Universidad de Rice (Moral, 2024): 2 × ∛1000 = 20.
La curva amarilla muestra la densidad estimada de dicha distribución empírica, mientras que la curva roja representa la densidad de la distribución bootstrap teórica de la estadística q, basada en seis variables normalmente distribuidas generadas con la misma estructura correlacional que las originales.
La curva verde corresponde a una distribución chi-cuadrado estándar con seis grados de libertad. La línea vertical amarilla indica el valor observado de la estadística q; la línea roja, el valor crítico (cuantil de orden .95) de la distribución bootstrap teórica; y la línea verde, el valor crítico correspondiente a la distribución chi-cuadrado estándar con seis grados de libertad.
La Figura 1 permite apreciar que el valor de la estadística de contraste queda a la izquierda de los valores críticos. Esto indica que se halla en la región de aceptación de la hipótesis nula de normalidad multivariante. La distribución bootstrap empírica y normativa son muy parecidas y se alejan de la distribución chi-cuadrado estándar por unos picos más bajos, unos hombros más planos y unas colas derechas más pesadas, ya que siguen una distribución chi-cuadrado generalizada.
Al no cumplirse el supuesto de independencia serial (con rezagos de 2 a 10), la distribución bootstrap normativa representa la alternativa más adecuada para calcular la probabilidad y tomar decisiones respecto a la hipótesis nula. Sobre todo, si se considera la semejanza entre los perfiles de la distribución muestral generada por bootstrap tanto desde su forma paramétrica (distribución muestral normativa) como no paramétrica (distribución muestral empírica), y la discrepancia de estos perfiles con el de la distribución chi-cuadrado estándar en que se basa la aproximación.
Figura 1
Prueba Q multivariante: histograma y densidades bootstrap vs. chi-cuadrado (con valor q y críticos)
Prueba QSF desde la aproximación chi-cuadrado y el enfoque bootstrap
En el Apéndice 2, se presenta el script para ejecutar la prueba Q de normalidad multivariante de Moral (2023) usando los valores estandarizados (Rotston, 1993) desde la prueba de normalidad univariante de Shapiro y Francia (1972). Se aplica a la misma muestra.
Tamaño muestral: n = 36.
Número de variables: k = 6.
Media de las correlaciones entre las variables: m(R) = .7127.
Nivel de significancia: α = .05 (aproximación chi-cuadrado) y .1 (enfoque bootstrap).
Parámetros comunes para la estandarización de las 63 variables creadas por combinación lineal de las 6 variables originales:
Valor esperado de la variable aleatoria ln(1-W’):
u = ln(ln(n)) – ln(n) = ln(ln(36)) – ln(36) = -2.3072.
μ = 1.0521 × u – 1.2725 = 1.0521 × -2.3072 – 1.2725 = -3.6999.
Desviación estándar de la variable aleatoria ln(1-W’):
v = ln(ln(n)) + 2 / ln(n) = ln(ln(36)) + 2 / ln(36) = 1.8345.
σ =-0.26758 × v + 1.0308 =-0.26758 × 1.8345 + 1.0308 = 0.5399.
En la Tabla 4, se puede observar que la hipótesis nula de normalidad univariante se sostiene en las 63 combinaciones lineales por la prueba de Shapiro y Francia (Royston, 1993) con un nivel de significancia del 5%.
Tabla 4
Prueba de normalidadunivariante de Shapiro y Francia con la estandarización de Royston
Número de combinaciones entre las variables originales: nc = 63.
Número de valores z anulados por ser negativos: a = 55.
Grados de libertad: gl = nc = 63.
Estadística de contraste: Q_SF = ∑(z’)² = 0.9628.
Valor de probabilidad y valor crítico desde una distribución chi-cuadrado estándar:
Valor crítico aproximado correspondiente a una distribución chi-cuadrado estándar con 63 grados de libertad: .95χ²[63] = 82.5287. Dado que q’ = 0.9628 < .95χ²[63] = 82.5287, se mantiene la hipótesis nula de normalidad multivariante con un nivel de significancia del 5 %.
Valor de probabilidad aproximado para q’: P(χ²[63] ≥ q’ = 0.9628) = 1. Dado que p = 1 > α = .05, se mantiene la hipótesis nula de normalidad multivariante con un nivel de significancia del 5 %.
Potencia estadística desde la aproximación chi-2 para un nivel de significancia de .05: phi = .0603.
Si no restara el número de valores anulados, igualmente se mantendría la hipótesis nula (q’ = 0.9628 < .95χ²[8] = 15.5073, p = .9985 > .05) con una potencia de ϕ = .0851.
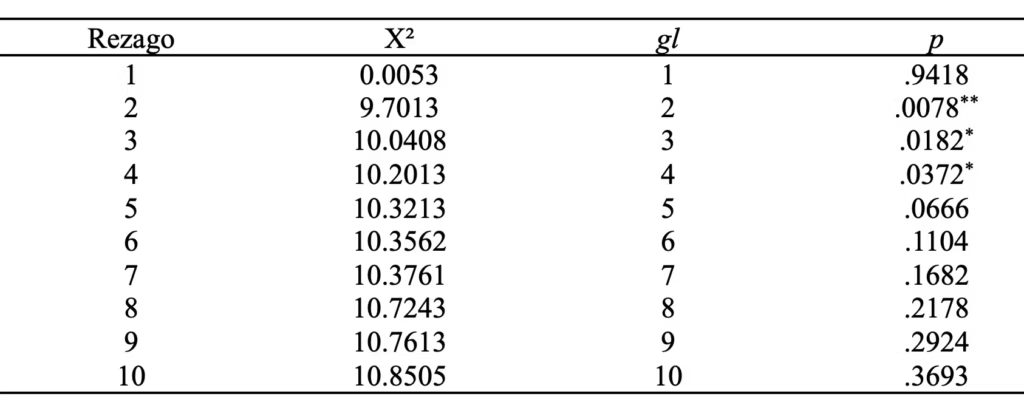
En la secuencia de valores de W’ logarítmicamente transformados, estandarizados y truncados (a 0 cuando son negativos), usados para calcular la estadística de contraste q’(z_w), no hay dependencia serial con base en la prueba de rachas de Wald y Wolfowitz (1943) y en la prueba de Ljung y Box (1978) con regazo de 1. No obstante, esta última prueba sí revela dependencia serial con rezagos de 2 a 4. El rezago máximo se establece por la regla general para series no estacionarias: min(10, n/5) = min(10, 63/5) = 10 (Chan et al., 2022). Véase Tabla 5.
Runs test for randomness. data: z_w: runs = 35, m = 32, n = 31, p = .5275 > .05.
Tabla 5
Prueba de Ljung y Box de independencia serial en la secuencia de valores z_w
Distribución bootstrap (con correlación conservada) empírica o desde la muestral original:
Número de muestras bootstrap: B = 1000.
Valor de probabilidad bootstrap a la cola derecha en la distribución bootstrap empírica:pBoot = 1. Dado que pBoot = 1 > α = .05, se mantiene la hipótesis nula de normalidad multivariante con un nivel de significancia del 5 %.
Distribución bootstrap normativa (con correlación conservada):
Número de muestras bootstrap: B = 1000.
Valor de la estadística q’ en la distribución bootstrap normativa: q’mvn_boot = 29.62837.
Media de la distribución bootstrap normativa: m(mvn_boot_dist) = 178.6752.
Mediana de la distribución bootstrap normativa: mdn(mvn_boot_dist) = 154.248.
Probabilidad de que la distribución bootstrap empírica esté centrada en la mediana de la distribución bootstrap normativa: p2 colas = .328 > α = .1. Consecuentemente, se mantiene hipótesis nula de medianas equivalentes entre las distribuciones bootstrap normativa y empírica en un contraste a dos colas con un nivel de significancia de 0.1.
Valor crítico bootstrap o cuantil de orden .90 de la distribución bootstrap normativa: q’mvn_crit = 321.4358. Dado que q’ = 0.9628 < q’mvn_crit = 321.4358, se mantiene la hipótesis nula de normalidad multivariante con un nivel de significancia del 10%.
Valor de probabilidad a la cola derecha en la distribución bootstrap normativa:pmvm_boot = 1 > α = .1. Consecuentemente, se mantiene la hipótesis nula de normalidad multivariante con un nivel de significancia del 10%.
Potencia estadística bootstrap para un nivel de significancia de .9: phi_boot = .018.
Con un nivel de significancia de .05, la conclusión sería la misma. El valor crítico bootstrap o cuantil de orden .95 de la distribución bootstrap normativa (q_crit) es 418.1868. Así, q’ = 0.9628 < q’mvn_crit = 379.4902. El valor de probabilidad a la cola derecha en la distribución bootstrap normativa (p_mvn_boot) es 1 > α = .05. La potencia estadística bootstrap para un nivel de significancia de .05 (phi_boot) es muy baja (ϕ = .006 < 0.2).
La Figura 2 permite ver que el valor de la estadística de contraste q‘ queda a la izquierda del valor crítico (cuantil de orden .95) de la distribución chi-cuadrado estándar con 63 grados de libertad y el valor crítico (cuantil de orden .90) de la distribución bootstrap normativa. Esto indica que está en la región de aceptación de la hipótesis nula de normalidad multivariante.
Además, se puede apreciar que la distribución muestral de la estadística q’ generada por bootstrap no paramétrico (desde los datos originales) o paramétrico (vectores gaussianos con una estructura correlacional equivalente a la de los datos originales) es muy semejante y se aleja de la distribución chi-cuadrado estándar con 63 grados de libertad por un pico más bajo, unos hombros más anchos y una cola derecha más pesada, ya que sigue una distribución chi-cuadrado generalizada. De ahí que se especifica un orden cuantil de .9 para la distribución bootstrap normativa, a fin de compensar su cola derecha muy pesada, y se omitió la corrección de eliminar el número de valores z’ truncados a cero cuando se calcula los grados de libertad de la distribución chi-cuadrado estándar (gl = 63 sin corrección versus 59 con corrección).
Figura 2
Histograma y densidades bootstrap y chi cuadrado para la prueba Q multivariante
Prueba H
Al contrastarse la normalidad multivariante mediante la prueba H de Royston, también se mantuvo la hipótesis nula de normalidad (H = 4.7374, p = .5038 > α = .05) con una potencia estadística baja para un nivel de significancia de .05 (phi = .3328 < .5). En el Apéndice 3, se muestra el guion de R para ejecutar estos cálculos.
La potencia relativa de la prueba H, en comparación con la aproximación chi-cuadrado (H/Q_aprox_chi2) del estadístico QSW, fue de 4.3793 (α = .05), y respecto al enfoque bootstrap (H/Q_boot), fue de 16.978 (α = .10). Dado que se mantiene la hipótesis nula de normalidad multivariante con valores de potencia son menores que .5, estos valores indican un mejor desempeño de la prueba Q.
De manera similar, la potencia relativa de la prueba H frente a la aproximación chi-cuadrado del estadístico QSF fue de 5.516 (α = .05), y frente al enfoque bootstrap, de 25.467 (α = .10). Esto también sugiere un mejor desempeño de la prueba Q. Por el contrario, en caso de que se rechace la hipótesis nula con valores de potencia mayores que .5, estos resultados implicarían una mayor potencia estadística de la prueba H.
Discusión
Se formuló como objetivo del estudio crear un script para el programa R e ilustrar su uso. A efectos prácticos se desarrollan tres scripts.
El primero para aplicar la prueba Q calculada con las estadísticas W de Shapiro y Wilk (Royston, 1992) desde su aproximación chi-cuadrado (QSWa) y el enfoque bootstrap (QSWb).
El segundo para aplicar la prueba Q calculada con las estadísticas W’ de Shapiro y Francia (Royston, 1993) desde su aproximación chi-cuadrado (QSFa) y el enfoque bootstrap (QSFb).
El tercero para aplicar la prueba H (Royston, 1983). Los tres scripts incluyen el cálculo del valor crítico, valor de probabilidad y potencia estadística. Adicionalmente, se calcula la potencia relativa de H con respecto a las dos variantes de la prueba Q (aproximación chi-cuadrado para una α =.05 o enfoque bootstrap para una α = .10). El ejemplo consistió en ejecutar los tres scripts en una muestra de 36 observaciones en seis variables (correlacionadas) que integran una escala unidimensional.
Las variables tienen un rango discreto de 1 a 10 y su suma define la puntuación en una escala total que sigue una distribución normal, lo que corroboran los datos muestrales (W = .9532, z = 1.1172, p = .132 y W’ = .9563, z’ = 1.0538, p = .146).
Cabe señalar que la prueba QSW está limitada a muestras de entre 3 y 2000 datos debido a la estandarización propuesta por Royston (1992), mientras que el estadístico QSF puede aplicarse a muestras de entre 5 y 5000 datos (Royston, 1993). Aunque el cálculo de QSW resulta más laborioso, su versión original —basada en la aproximación chi-cuadrado— ofrece un rendimiento ligeramente superior al de QSF en contextos con muestras pequeñas y pocas variables (Moral, 2023).
La expectativa era de cumplimiento del supuesto de normalidad multivariante de forma coherente por las cinco pruebas. De acuerdo con la expectativa, la hipótesis nula de normalidad multivariante se sostuvo con las cinco pruebas. La potencia fue baja con la prueba H de Royston (1983) y muy baja con las cuatro variantes de la prueba Q, más baja con las variantes bootstrap que con la aproximación chi-cuadrado.
Dado que se incumple el supuesto de independencia serial, por la prueba de Ljung y Box (1978) con rezagos mayores que uno, la distribución bootstrap normativa es una opción más adecuada para calcular la probabilidad y tomar decisiones respecto a la hipótesis nula que la distribución chi-cuadrado estándar. Además, esta elección se ve reforzado por la semejanza entre los perfiles de la distribución muestral generada por muestreo repetitivo, ya sea usando la distribución normal multivariante (bootstrap paramétrico) o la muestra original (bootstrap no paramétrico), y el alejamiento de ambos perfiles del contorno de la distribución chi-cuadrado estándar en que se basa cálculo aproximado de la prueba Q. Esta aproximación se fundamenta en que la distribución muestral (teórica) de la suma de cuadrados de variables independientes normalmente distribuidas es una distribución chi-cuadrado estándar con tantos grados de libertad como variables sumadas (Moral, 2023).
Los perfiles generados son menos apuntados y con una cola derecha más pesada que la distribución chi-cuadrado estándar, ya que corresponden a una distribución chi-cuadrado generalizada que siguen las sumas de cuadrados de variables correlacionadas (Ferrari, 2019). Este perfil motiva a que no se elimina el número de valores z truncados a cero (gl = 63 sin corrección versus 10 con corrección en QSWa y 63 versus 8 en QSFa) cuando se calculan los grados de libertad de la distribución chi-cuadrado estándar. De este modo, se logra que el punto crítico esté más a la derecha. Esta también es la razón por la cual se modifica el orden cuantil de .95 a .9 cuando se calcula el valor crítico de la distribución bootstrap normativa. Así, se compensa su cola derecha muy pesada, quedando el punto crítico más a la izquierda. Estos cambios intentan aproximar las conclusiones con ambas distribuciones (chi-cuadrado estándar y chi-cuadrado generalizada).
La proximidad de las dos distribuciones bootstrap (normativa y empírica) no sólo se puede valorar visualmente con la gráfica que proporciona el script, sino por medio del contraste de la equivalencia de sus medianas, lo que positivamente se corrobora. La distribución bootstrap normativa se usa en lugar de la distribución chi-cuadrado estándar para el cálculo del valor de probabilidad y permite obtener el punto de corte para definir la región de rechazo y calcular la potencia en la distribución bootstrap empírica. Cabe señalar que una de las utilidades más importantes del método bootstrap es acceder a la distribución muestral de un estimador sin asumir supuestos (Rousselet et al., 2023).
El script también calcula el valor de probabilidad utilizando la distribución muestral empírica obtenida mediante bootstrap no paramétrico. Este enfoque es comúnmente empleado cuando la hipótesis nula se refiere a una medida descriptiva o de asociación cuya distribución muestral es desconocida. En cambio, si la hipótesis nula se basa en una distribución conocida, es preferible aplicar el bootstrap paramétrico bajo esa hipótesis (Rousselet et al., 2023).
Dado que la muestra fue diseñada para cumplir con el supuesto, las pruebas funcionan correctamente al sostener dicha hipótesis. En relación con este hecho, cabe señalar que el presente trabajo metodológico presenta la limitación de no evaluar el rendimiento de la prueba Q en términos de proporción de aciertos y potencia estadística, considerando distribuciones multivariantes, tanto normales como no normales.
En este sentido, sería importante tener en cuenta factores como el tamaño de la muestra, el número de variables y las correlaciones entre ellas (Jobst et al., 2023; Norton & Strube, 2001), así como comparar las cuatro versiones de la prueba Q entre sí, con la prueba H, e incluso con otras pruebas de normalidad multivariante, como la de Henze y Zirkler (1990), la cual está implementada en R (Korkmaz, 2025). Se sugiere que futuras investigaciones mediante simulación aborden estos aspectos.
Conclusiones
La distribución muestral de la estadística Q relevada por los métodos bootstrap (paramétrico y no paramétrico) corresponde a una distribución chi-cuadrado generalizada que se aleja por menor apuntamiento y más asimetría positiva de una distribución chi-cuadrado estándar. El ejemplo indica que la variante bootstrap de la prueba Q funciona mejor cuando se calcula con las estadísticas W’ de Shapiro y Francia.
Este enfoque es conservador con la hipótesis nula de normalidad multivariante en comparación con la prueba H en las variantes basadas en la aproximación chi-cuadrado de la prueba Q, por lo que debe incrementarse el nivel de significancia a 10 %. Considerando la distribución muestral de la estadística Q que revela el enfoque bootstrap, no se debe restar el número de estadísticas W o W’ estandarizadas y truncadas a 0, cuando se calculan los grados de libertad de la variante basada en la aproximación chi-cuadrado de la prueba Q.
Sugerencias
Se recomienda el uso del presente script con las cuatro variantes de la prueba Q y la prueba H. En caso de sostenerse el supuesto de independencia serial, valorado por las pruebas de contraste y las gráficas, la aproximación chi-cuadrado es opción menos conservadora con la hipótesis nula. En caso de incumplimiento, el enfoque bootstrap es teóricamente más adecuado, siendo mejor opción su cálculo con las estadísticas W’ de Shapiro y Francia y un nivel de significancia del 10 %. La convergencia de resultados con la prueba H refuerza la conclusión extraída.
Finalmente, se presenta una lista de buenas prácticas para aplicar la prueba Q y H:
1. Ejecutar el script con las cuatro variantes de la prueba Q y con la prueba H, para obtener una evaluación más completa y robusta del ajuste a la normalidad multivariante.
2. Elegir el enfoque bootstrap de la prueba Q cuando no se cumple el supuesto de independencia serial, ya que ofrece una distribución muestral más adecuada para este contexto.
3. Utilizar las estadísticas W’ de Shapiro y Francia al implementar la variante bootstrap, dado su mejor rendimiento en muestras pequeñas.
4. Ajustar el nivel de significancia al 10 % en el enfoque bootstrap, considerando su naturaleza conservadora frente a la hipótesis nula de normalidad multivariante.
5. Evitar restar el número de estadísticas truncadas a cero al calcular los grados de libertad en la variante basada en la aproximación chi-cuadrado de la prueba Q.
6. Emplear la aproximación chi-cuadrado de la prueba Q únicamente cuando se confirme la independencia serial, mediante pruebas de contraste y análisis gráfico.
Agradecimientos
El autor agradece las observaciones del editor de la revista PsicologiaCientifica.com y los aportes recibidos durante la evaluación editorial, los cuales contribuyeron significativamente a mejorar la calidad del manuscrito.
Financiamiento
Este estudio fue financiado con recursos propios del autor.
Conflictos de intereses
El autor declara que no existen conflictos de interés relacionados con el estudio presentado en este artículo.
Uso de herramientas de inteligencia artificial
El autor declara que no ha utilizado herramientas de inteligencia artificial para la generación o el análisis de datos, el diseño de gráficos ni cualquier otro aspecto técnico del estudio.
Anis, W., Kuntoro, S. M., & Melaniani, S. (2021). Difference of power test and type II error (β) on Mardia MVN test, Henze-Zirkler’s MVN test, and Royston’s MVN test using multivariate data analysis. Jurnal Biometrika dan Kependudukan, 10(2), 153-161. https://doi.org/10.20473/jbk.v10i2.2021.153-161
Braun, W. J., & Murdoch, D. J. (2021). A first course in statistical programming with R (3rd ed.). Cambridge University Press.
Chan, K. C. G., Han, J., Kennedy, A. P., & Yam, S. C. P. (2022). Testing network autocorrelation without replicates. PLOS ONE, 17(11), e0275532. https://doi.org/10.1371/journal.pone.0275532
Chen, T., & Lumley, T. (2019). Numerical evaluation of methods approximating the distribution of a large quadratic form in normal variables. Computational Statistics & Data Analysis, 139, 75-81. https://doi.org/10.1016/j.csda.2019.05.002
Egbert, J., & Plonsky, L. (2021). bootstrapping techniques. En M. Paquot & S. T. Gries (Eds.), A practical handbook of corpus linguistics (pp. 593-610). Springer. https://doi.org/10.1007/978-3-030-46216-1_24
Ferrari, A. (2019). A note on sum and difference of correlated chi-squared variables. arXiv. https://doi.org/10.48550/arXiv.1906.09982
Giorgi, F. M., Ceraolo, C., & Mercatelli, D. (2022). The R language: An engine for bioinformatics and data science. Life, 12(5), 648. https://doi.org/10.3390/life12050648
Goetze, F., & Tikhomirov, A. (2002). Asymptotic distribution of quadratic forms and applications. Journal of Theoretical Probability, 15(2), 423-475. https://doi.org/10.1023/A:1014867011101
Gross, J., & Ligges, U. (2022). nortest: Tests for normality (Version 1.0-4) [Computer software]. CRAN. https://cran.r-project.org/web/packages/nortest/nortest.pdf
Henze, N., & Zirkler, B. (1990). A class of invariant consistent tests for multivariate normality. Communications in Statistics – Theory and Methods, 19(10), 3595-3617. https://doi.org/10.1080/03610929008830400
Hyndman, R. J. & Athanasopoulos, G. (2021). Forecasting: principle and practice (3rd ed.). Melbourne, Australia: OTexts.
Jobst, L. J., Bader, M., & Moshagen, M. (2023). A tutorial on assessing statistical power and determining sample size for structural equation models. Psychological Methods, 28(1), 207-221. https://doi.org/10.1037/met0000423
Khatun, N. (2021). Applications of normality test in statistical analysis. Open Journal of Statistics, 11(1), 113-122. https://doi.org/10.4236/ojs.2021.111006
Korkmaz, S. (2025). MVN: Multivariate normality tests (Version 5.0) [Computer software]. CRAN. https://cran.r-project.org/web/packages/MVN/MVN.pdf
Ljung, G. M., & Box, G. E. P. (1978). On a measure of a lack of fit in time series models. Biometrika, 65(2), 297-303. https://doi.org/10.1093/biomet/65.2.297
Marchev, A., Jr., & Marchev, V. (2024). Automated algorithm for multivariate data synthesis with Cholesky decomposition. En Proceedings of the 7th International Conference on Algorithms, Computing and Systems (pp. 1-6). Association for Computing Machinery. https://doi.org/10.1145/3631908.3631909
Mardia, K. V. (1980). Tests of univariate and multivariate normality. En P. R. Krishnaiah (Ed.), Handbook of statistics 1: Analysis of variance (pp. 279-320). North-Holland. https://doi.org/10.1016/S0169-7161(80)01011-5
Moral, J. (2023). Proposal and pilot study: A generalization of the W or W′ statistic for multivariate normality. Open Journal of Statistics, 13(1), 119-169. https://doi.org/10.4236/ojs.2023.131008
Moral, J. (2024). Rice university rule to determine the number of bins. Open Journal of Statistics, 14(1), 119-149. https://doi.org/10.4236/ojs.2024.141006
R Core Team. (2025a). R: A language and environment for statistical computing [Computer software]. R Foundation for Statistical Computing. https://www.r-project.org/
R Core Team. (2025b). The R Stats package (Version 4.6.0) [Computer software]. https://stat.ethz.ch/R-manual/R-devel/library/stats/html/00Index.html
Ripley, B., Venables, B., Bates, D. M., Hornik, K., Gebhardt, A., & Firth, D. (2025). MASS: Support functions and datasets for Venables and Ripley’s MASS (Version 7.3-65) [Computer software]. https://doi.org/10.32614/CRAN.package.MASS
Rolke, W., & Gongora, C. G. (2021). A chi-square goodness-of-fit test for continuous distributions against a known alternative. Computational Statistics, 36(3), 1885-1900. https://doi.org/10.1007/s00180-020-00997-x
Rousselet, G., Pernet, C. R., & Wilcox, R. R. (2023). An introduction to the bootstrap: A versatile method to make inferences by using data-driven simulations. Meta-Psychology, 7, 1-38. https://doi.org/10.15626/MP.2019.2058
Royston, J. P. (1983). Some techniques for assessing multivariate normality based on the Shapiro-Wilk W. Journal of the Royal Statistical Society. Series C (Applied Statistics), 32(2), 121-133. https://doi.org/10.2307/2347291
Royston, J. P. (1992). Approximating the Shapiro-Wilk W-test for non-normality. Statistics and Computing, 2(3), 117-119. https://doi.org/10.1007/BF01891203
Royston, J. P. (1993). A toolkit for testing non-normality in complete and censored samples. Journal of the Royal Statistical Society. Series D (The Statistician), 42(1), 37-43. https://doi.org/10.2307/2348109
Schmal, W. B. (2024). Quantitative tools for time series analysis in natural language processing: A practitioners guide. arXiv. https://doi.org/10.48550/arXiv.2404.18499
Schwert, G. W. (1989). Why does stock market volatility change over time? The Journal of Finance, 44(5), 1115-1153. https://doi.org/10.1111/j.1540-6261.1989.tb02647.x
Shapiro, S. S., & Francia, R. S. (1972). An approximate analysis of variance test for normality. Journal of the American Statistical Association, 67(337), 215-216. https://doi.org/10.1080/01621459.1972.10481232
Shapiro, S. S., & Wilk, M. B. (1965). An analysis of variance test for normality (complete samples). Biometrika, 52(3-4), 591-611. https://doi.org/10.2307/2333709
Signorell, A. (2025). DescTools: Tools for descriptive statistics (Version 0.99.6) [Computer software]. https://doi.org/10.32614/CRAN.package.DescTools
Wald, A., & Wolfowitz, J. (1943). An exact test for randomness in the non-parametric case based on serial correlation. The Annals of Mathematical Statistics, 14(4), 378-388. https://doi.org/10.1214/aoms/1177731358
Apéndices
# Prueba Q de normalidad multivariante desde las estadísticas W de normalidad univariante de Shapiro y Wilk (1965) estandarizadas por el método de Royston (1992) para muestras de tamaño de 12 a 2000 con k mediciones.
library(DescTools)
# Cargar cuatro paquetes requeridos.
library(nortest)
library(MASS)
library(stats)
# Se recomienda la última versión de R.
# El paquete ‘nortest’ no especifica una versión mínima clara; la versión actual 1.0-4 funciona en R 4.4 y versiones anteriores.
# La versión 7.3-65 del paquete ‘MASS’ requiere R (≥ 4.4.0).
# La versión 0.99.60 del paquete ‘DescTools’ requiere R (≥ 4.2.0).
# El paquete ‘stats’ es un paquete base de R y se actualiza junto con R.
# Si no se tienen los paquetes, seleccionar espejo más próximo: chooseCRANmirror()
# install.packages(«nortest»)
# install.packages(«MASS»)
# install.packages(«DescTools»)
# Lista de variables originales.
x1 <- c(7, 6, 4, 5, 3, 9, 5, 3, 3, 5, 5, 6, 5, 9, 6, 10, 8, 7, 7, 3, 6, 5, 8, 7, 1, 4, 7, 2, 6, 4, 4, 8, 6, 2, 1, 4)
x2 <- c(6, 7, 4, 4, 5, 7, 6, 5, 3, 4, 7, 5, 1, 8, 5, 10, 8, 6, 8, 2, 4, 2, 7, 9, 3, 3, 5, 3, 5, 4, 6, 7, 6, 6, 1, 4)
x3 <- c(7, 9, 5, 5, 6, 9, 5, 5, 6, 2, 6, 7, 2, 10, 6, 9, 9, 3, 8, 2, 4, 4, 9, 6, 2, 3, 7, 4, 3, 6, 4, 7, 6, 5, 1, 4)
x4 <- c(8, 7, 6, 7, 3, 9, 4, 2, 3, 4, 5, 5, 1, 10, 6, 8, 9, 2, 6, 2, 6, 4, 7, 7, 5, 5, 7, 5, 5, 4, 6, 6, 8, 3, 1, 4)
x5 <- c(6, 7, 4, 6, 3, 6, 8, 2, 4, 4, 8, 8, 4, 10, 6, 8, 7, 4, 6, 2, 2, 4, 9, 9, 3, 4, 4, 6, 4, 6, 6, 7, 7, 3, 1, 3)
x6 <- c(8, 6, 4, 7, 5, 9, 8, 3, 5, 7, 6, 6, 5, 10, 4, 10, 9, 4, 6, 3, 6, 2, 8, 8, 5, 3, 7, 7, 4, 5, 6, 7, 5, 3, 2, 4)
datos_orig <- data.frame(x1, x2, x3, x4, x5, x6)
# Tamaño muestral, número de variables y nivel de significancia.
n <- length(x1)
cat(«\nTamaño muestral: n =», n, «.\n»)
k <- length(datos_orig)
cat(«Número de variables en la muestra original: k =», k, «.\n»)
R <- cor(datos_orig)
m_R <- mean(R[lower.tri(R)])
cat(«Media de las correlaciones entre las variables: m(R) =», round(m_R, 4), «.\n»)
alpha <- 0.05
# Generar las sumas de las combinaciones sin repetición entre las variables.
generar_combinaciones_lineales <- function(df) {
lista_combinaciones <- list()
k <- ncol(df)
# Agregar las variables originales (combinaciones de tamaño 1).
for (i in 1:k) {
lista_combinaciones[[paste0(«c», i)]] <- df[, i]}
# Agregar combinaciones de tamaño 2 hasta k.
contador <- k
for (i in 2:k) {
combinaciones <- combn(k, i)
for (j in 1:ncol(combinaciones)) {
idx <- combinaciones[, j]
contador <- contador + 1
lista_combinaciones[[paste0(«c», contador)]] <- rowSums(df[, idx])}}
return(lista_combinaciones)}
lista_x <- generar_combinaciones_lineales(datos_orig)
# Cálculo de los parámetros comunes para la estandarización para tamaños muestrales de 12 a 2000.
mu <- 0.0038915 * log(n)^3 – 0.083751 * log(n)^2 – 0.31082 * log(n) – 1.5861
sigma <- exp(0.0030302 * log(n)^2 – 0.082676 * log(n) – 0.4803)
cat(«\nParámetros comunes para la estandarización de las», 2^k – 1, «variables creadas por combinación lineal de las», k, «variables originales:\n»)
cat(«\nValor esperado de ln(1-W): μ =», mu, «.\n»)
cat(«Desviación estándar de ln(1-W): σ =», sigma, «.\n»)
# Aplicar prueba y guardar en tabla.
resultados <- lapply(names(lista_x), function(nombre) {
datos <- lista_x[[nombre]]
resultado <- shapiro.test(datos)
z <- (log(1 – resultado$statistic) – mu) / sigma
p <- 1 – pnorm(z)
Normalidad <- if (resultado$p.value < alpha) «No» else «Si»
return(c(nombre, round(resultado$statistic, 4), round(z, 4), round(p, 4), Normalidad))
})
# Convertir a marco de datos (data frame).
tabla_resultados <- as.data.frame(do.call(rbind, resultados), stringsAsFactors = FALSE)
colnames(tabla_resultados) <- c(«Variable», «W», «z», «p», «Normalidad»)
# Mostrar tabla.
cat(«\nTabla. Prueba de normalidad univariante de Shapiro y Wilk con la estandarización de Royston para las», 2^k-1, «combinaciones lineales de variables\n»)
print(tabla_resultados, row.names = FALSE)
cat(«\nNota. Variable = suma de la correspondiente combinación sin repetición de las variables; W = estadística W de Shapiro y Wilk (1965); z = valor estandarizado de W mediante la estandarización de Royston (1992); p = valor de probabilidad a la cola derecha de una distribución normal estándar; y Normalidad univariante: Sí = p ≥ α =», alpha, «, se mantiene la hipótesis nula de normalidad; y No = p <«, alpha, «, se rechaza.\n»)
# Función para calcular q.
calcular_q <- function(lista_datos) {
z_vals <- sapply(lista_datos, function(x) {
resultado_sw <- shapiro.test(x)
z <- (log(1 – resultado_sw$statistic) – mu) / sigma
return(as.numeric(z))
})
z_values <- ifelse(z_vals < 0, 0, z_vals)
a <- sum(z_vals < 0)
q <- sum(z_values^2)
return(list(q = q, a = a, z_vals = z_vals, z_values = z_values))
}
# Valor de probabilidad con la distribución chi-cuadrado estándar.
resultado_q <- calcular_q(lista_x)
q_stat <- resultado_q$q
a <- resultado_q$a
nc <- length(lista_x)
# gl_chisq <- nc – a # con corrección por valores truncados.
gl_chisq <- nc # sin corrección por valores truncados.
q_crit <- qchisq(1 – alpha, df = gl_chisq)
p_value <- pchisq(q_stat, df = gl_chisq, lower.tail = FALSE)
power <- 1 – pchisq(q_crit, df = gl_chisq, ncp = q_stat, lower.tail = TRUE)
# Resultados para QSWa.
cat(«\nNúmero de combinaciones entre las», k, «variables originales: nc =», nc, «.\n»)
cat(«Número de valores z anulados por ser negativos: a =», a, «.\n»)
cat(«Grados de libertad: gl =», gl_chisq, «.\n»)
cat(«\nEstadística de contraste: q =», round(q_stat, 4), «.\n»)
cat(«\nValor de probabilidad y valor crítico desde una distribución chi-cuadrado estándar:\n»)
cat(«\nValor crítico o cuantil de orden», 1 – alpha,» de una distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad: 1-αχ²[gl] =», round(q_crit, 4), «.\n»)
if (q_stat <= q_crit) {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», alpha, «con base en el valor crítico desde la aproximación chi-cuadrado.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», alpha, «con base en el valor crítico desde la aproximación chi-cuadrado.\n»)}
cat(«Valor de probabilidad a la cola derecha en una distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad: p =», round(p_value, 4), «.\n»)
if (p_value < alpha) {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», alpha, «con base en el valor de probabilidad desde la aproximación chi-cuadrado.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», alpha, «con base en el valor de probabilidad desde la aproximación chi-cuadrado.\n»)}
cat(«Potencia estadística desde la aproximación chi-cuadrado para un nivel de significancia de», alpha, «: phi =», power, «.\n»)
cat(«Si se rechaza la hipótesis nula, la potencia estadística debería ser superior a 0.5, preferiblemente mayor que 0.8; mientras que, si no se rechaza la hipótesis nula, debería ser inferior a 0.5, preferiblemente menor que 0.2. En otro caso, el resultado es contradictorio o cuestionable.\n»)
# Contraste de la independencia serial en los valores z.
z_w <- resultado_q$z_values
cat(«\nContraste de la independencia entre los», nc, «valores de W logarítmicamente transformados, estandarizados y truncados (a 0 cuando son negativos), usados para calcular q(z_w).\n»)
RunsTest(z_w, y = NULL, alternative = «two.sided», exact = TRUE, correct = FALSE)
lag_max <- min(10, round(nc/5, 0)) # Regla de Hyndman y Athanasopoulos (2021) para determinar el rezago máximo en series temporales no estacionarias.
# lag_max <- 12 * (n/100)^0.25 # Regla de Schwert para determinar el rezago máximo.
ljung_box_test <- lapply(1: lag_max, function(k) Box.test(z_w, lag = k, type = «Ljung-Box», fitdf = 0))
ljung_box_tabla <- data.frame(
Rezago = 1:lag_max,
Chi_cuadrado = sapply(ljung_box_test, function(x) round(x$statistic, 4)),
gl = sapply(ljung_box_test, function(x) round(x$parameter, 4)),
p = sapply(ljung_box_test, function(x) round(x$p.value, 4))
)
cat(«\nTabla. Prueba de Ljung y Box de independencia serial en la secuencia de valores z_w\n»)
print(ljung_box_tabla)
cat(«\nNota. Rezago = intervalo entre los valores que conforman los pares de datos en la autocorrelación, X² = estadística de contraste, gl = grados de libertad y p = probabilidad a la cola derecha en una distribución chi-cuadrado estándar.\n»)
# Función bootstrap preservando las correlaciones de la muestra original.
resample_preserving_corr <- function(df, B) {
replicate(B, {
df[sample(1:nrow(df), size = nrow(df), replace = TRUE), ]
}, simplify = FALSE)
}
# Función para generar lista de combinaciones.
generar_lista_boot <- function(df) {
lista_boot <- setNames(as.list(df), paste0(«c», 1:ncol(df)))
for (i in 2:ncol(df)) {
combinaciones <- combn(ncol(df), i)
for (j in 1:ncol(combinaciones)) {
idx <- combinaciones[, j]
nombre <- paste0(«c», length(lista_boot) + 1)
lista_boot[[nombre]] <- rowSums(df[, idx])
}
}
return(lista_boot)}
# Simulaciones bootstrap preservando correlaciones.
set.seed(123) # Se fija una semilla para reproducibilidad
B <- 1000 # Número de remuestreos con reposición desde la muestra original
boot_datasets <- resample_preserving_corr(datos_orig, B)
q_boot_vals <- sapply(boot_datasets, function(df_boot) {
lista_boot <- generar_lista_boot(df_boot)
calcular_q(lista_boot)$q
})
# Valor de probabilidad en la distribución bootstrap empírica.
boot_p_value <- mean(q_boot_vals >= q_stat)
cat(«\nDistribución bootstrap (con correlación conservada) empírica o desde la muestral original:\n»)
cat(«Número de muestras bootstrap: B =», B, «.\n»)
cat(«Valor de probabilidad bootstrap a la cola derecha en la distribución bootstrap empírica: p_boot =», round(boot_p_value, 4), «.\n»)
if (boot_p_value < alpha) {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», alpha, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap empírica.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», alpha, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap empírica.\n»)}
# Generar una muestra normativa con probabilidades equiespaciadas y la transformación Cholesky de la matriz de correlaciones de la muestra original.
p <- seq(0.5 / n, 1 – 0.5 / n, length.out = n)
datos_indep <- matrix(NA, nrow = n, ncol = k)
set.seed(123) # Se fija una semilla para reproducibilidad
for (j in 1:k) {datos_indep[, j] <- qnorm(sample(p))}
mat_cor <- cor(datos_orig)
mat_chol <- chol(mat_cor)
datos_normativos <- datos_indep %*% mat_chol
# Definir las variables-suma de todas las posibles combinaciones entre las variables originales.
lista_x_norm <- generar_combinaciones_lineales(datos_normativos)
result_q_norm <- calcular_q(lista_x_norm)
# Generar distribución bootstrap normativa con remuestreo de filas completas.
B <- 1000 # Número de remuestreos con reposición desde la muestra normativa
q_boot_norm_vals <- replicate(B, {
indices <- sample(1:n, size = n, replace = TRUE)
muestra_boot <- datos_normativos[indices, ]
lista_boot_n <- generar_combinaciones_lineales(muestra_boot)
calcular_q(lista_boot_n)$q
})
# Valor de probabilidad y valor crítico en la distribución bootstrap normativa.
boot_norm_p_value <- mean(q_boot_norm_vals >= q_stat)
# orden_cuantil <- 1-alpha # Sin compensación por cola derecha muy pesada.
orden_cuantil <- 1-2*alpha # Con compensación por cola derecha muy pesada.
valor_critico_boot_norm <- quantile(q_boot_norm_vals, probs = orden_cuantil)
cat(«\nDistribución bootstrap normativa (con correlación conservada):\n»)
cat(«Número de muestras bootstrap: B =», B, «.\n»)
cat(«Valor de la estadística q en la muestra normativa: q_mvn_boot =», result_q_norm$q, «.\n»)
cat(«Media de la distribución bootstrap normativa: m(mvn_boot_dist) =», mean(q_boot_norm_vals), «.\n»)
cat(«Mediana de la distribución bootstrap normativa: mdn(mvn_boot_dist) =», median(q_boot_norm_vals), «.\n»)
p_value_mdn_norm <- 2 * min(mean(q_boot_vals >= median(q_boot_norm_vals)), mean(q_boot_vals <= median(q_boot_norm_vals)))
cat(«Probabilidad de que la distribución bootstrap empírica esté centrada en la mediana de la distribución bootstrap normativa: p_2 colas =», round(p_value_mdn_norm, 4), «.\n»)
if (p_value_mdn_norm < alpha) {cat(«Se rechaza hipótesis nula de medianas equivalentes entre las distribuciones bootstrap normativa y empírica en un contraste a dos colas con un nivel de significancia de», 1-orden_cuantil, «.\n»)
} else {cat(«Se mantiene hipótesis nula de medianas equivalentes entre las distribuciones bootstrap normativa y empírica en un contraste a dos colas con un nivel de significancia de «, 1-orden_cuantil, «.\n»)}
cat(«Valor crítico bootstrap o cuantil de orden», orden_cuantil, «de la distribución bootstrap normativa: q_crit =», round(valor_critico_boot_norm, 4), «.\n»)
if (q_stat <= valor_critico_boot_norm) {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», 1-orden_cuantil, «con base en el valor crítico bootstrap (normativo).\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», 1- orden_cuantil, «con base en el valor crítico bootstrap (normativo).\n»)}
cat(«Valor de probabilidad a la cola derecha en la distribución bootstrap normativa: p_mvn_boot =», round(boot_norm_p_value, 4), «.\n»)
if (boot_norm_p_value < 1-orden_cuantil) {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», 1-orden_cuantil, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap normativa.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», 1-orden_cuantil, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap normativa.\n»)}
# Potencia bootstrap.
boot_power <- mean(q_boot_vals > valor_critico_boot_norm)
cat(«Potencia estadística bootstrap para un nivel de significancia de», 1-orden_cuantil, «: phi_boot =», boot_power, «.\n»)
# Representación de las distribuciones muestral bootstrap (empírica y normativa).
# Quitar los símbolos hashtag precedentes para grabar como un archivo JPG o TIFF
# jpeg(«Hist_curva_dens1.jpg», width = 1200, height = 900, units = «px», res = 300)
# tiff(«Hist_curva_dens1.tiff», width = 1200, height = 900, units = «px», res = 300)
# par(mar = c(4.5, 4.5, 0.5, 0.5), cex.axis = 0.8)
# Curva de densidad de la distribución chi-cuadrado estándar como gráfico base.
x_seq <- seq(0, max(qchisq(0.999, df = gl_chisq), max(q_boot_vals) +1, q_stat +1, max(q_boot_norm_vals) +1), length.out = 1000)
y_chi_sq <- dchisq(x_seq, df = gl_chisq)
plot(x_seq, y_chi_sq, type = «l», lwd = 3, col = «darkgreen»,
main = «», xlab = «Valores q», ylab = «Densidad»,
xlim = c(0, max(qchisq(0.999, df = gl_chisq), max(q_boot_vals) +1, q_stat +1, max(q_boot_norm_vals) +1)),
ylim = c(0, max(density(q_boot_vals)$y, y_chi_sq)))
# Histograma amarillo de la distribución bootstrap empírica de la estadística de contraste q.
hist(q_boot_vals, breaks = 20, freq = FALSE, col = rgb(1, 1, 0, 0.5), border = «yellow2», add = TRUE)
# Curva amarilla de densidad de la distribución bootstrap empírica de la estadística de contraste q.
lines(density(q_boot_vals), col = «yellow3», lwd = 2)
# Curva roja de densidad bootstrap normativa.
lines(density(q_boot_norm_vals), col = «red», lwd = 2)
# Línea vertical violeta para el valor de la estadística de contraste q observado.
abline(v = q_stat, col = «purple», lwd = 2, lty = 2)
# Línea vertical roja para el valor crítico normativo bootstrap.
abline(v = valor_critico_boot_norm, col = «red», lwd = 2, lty = 2)
# Línea vertical verde para el valor crítico de la distribución chi-cuadrado estándar.
abline(v = qchisq(1-alpha, df = gl_chisq), col = «darkgreen», lwd = 2, lty = 2)
# dev.off() # Quitar el símbolo hashtag precedente cuando se graba la figura.
cat(«\nFigura 1. Histograma (amarillo) con las curvas de densidad de las distribuciones bootstrap empírica (amarilla), bootstrap normativa (roja) y chi-cuadrado estándar (verde). El valor de la estadística de contraste q se representa mediante una línea vertical violeta (q =», q_stat, «), el valor crítico de la distribución bootstrap normativa mediante una línea vertical roja («, orden_cuantil, «_q_mvn_boot =», valor_critico_boot_norm, «) y el valor crítico de la distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad mediante una línea vertical verde («, 1-alpha, «χ²[«, gl_chisq, «] =», q_crit, «).\n»)
cat(«\nNota. Histograma amarillo (regla de la Universidad de Rice) = densidades de la distribución bootstrap empírica de la estadística de contraste q,
curva amarilla = curva de densidad de la distribución bootstrap empírica de la estadística de contraste q,
curva roja = curva de densidad de la distribución bootstrap normativa (generada desde variables normalmente distribuidas con la misma estructura correlacional que las originales) de la estadística de contraste q,
curva verde = distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad,
línea vertical violeta = valor de la estadística de contraste q en la muestra original,
línea vertical roja = valor crítico o cuantil de orden», orden_cuantil, «de la distribución bootstrap normativa,
línea vertical verde = valor crítico o cuantil de orden», 1-alpha, «de una distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad.\n»)
# Prueba Q’ de normalidad multivariante desde las estadísticas W’ de normalidad univariante de Shapiro y Francia (1972) estandarizadas por el método de Royston (1993) para muestras de tamaño de 5 a 5000 con k mediciones.
# Cargar cuatro paquetes requeridos.
library(nortest)
library(MASS)
library(DescTools)
library(stats)
# Lista de variables originales.
x1 <- c(7, 6, 4, 5, 3, 9, 5, 3, 3, 5, 5, 6, 5, 9, 6, 10, 8, 7, 7, 3, 6, 5, 8, 7, 1, 4, 7, 2, 6, 4, 4, 8, 6, 2, 1, 4)
x2 <- c(6, 7, 4, 4, 5, 7, 6, 5, 3, 4, 7, 5, 1, 8, 5, 10, 8, 6, 8, 2, 4, 2, 7, 9, 3, 3, 5, 3, 5, 4, 6, 7, 6, 6, 1, 4)
x3 <- c(7, 9, 5, 5, 6, 9, 5, 5, 6, 2, 6, 7, 2, 10, 6, 9, 9, 3, 8, 2, 4, 4, 9, 6, 2, 3, 7, 4, 3, 6, 4, 7, 6, 5, 1, 4)
x4 <- c(8, 7, 6, 7, 3, 9, 4, 2, 3, 4, 5, 5, 1, 10, 6, 8, 9, 2, 6, 2, 6, 4, 7, 7, 5, 5, 7, 5, 5, 4, 6, 6, 8, 3, 1, 4)
x5 <- c(6, 7, 4, 6, 3, 6, 8, 2, 4, 4, 8, 8, 4, 10, 6, 8, 7, 4, 6, 2, 2, 4, 9, 9, 3, 4, 4, 6, 4, 6, 6, 7, 7, 3, 1, 3)
x6 <- c(8, 6, 4, 7, 5, 9, 8, 3, 5, 7, 6, 6, 5, 10, 4, 10, 9, 4, 6, 3, 6, 2, 8, 8, 5, 3, 7, 7, 4, 5, 6, 7, 5, 3, 2, 4)
datos_orig <- data.frame(x1, x2, x3, x4, x5, x6)
# Tamaño muestral, número de variables y nivel de significancia.
n <- length(x1)
k <- length(datos_orig)
cat(«\nTamaño muestral: n =», n, «.\n»)
cat(«Número de variables en la muestra original: k =», k, «.\n»)
R <- cor(datos_orig)
m_R <- mean(R[lower.tri(R)])
cat(«Media de las correlaciones entre las variables: m(R) =», round(m_R, 4), «.\n»)
alpha <- 0.05
# Generar las sumas de las combinaciones sin repetición entre las variables.
generar_combinaciones_lineales <- function(df) {
lista_combinaciones <- list()
k <- ncol(df)
# Agregar las variables originales (combinaciones de tamaño 1).
for (i in 1:k) {
lista_combinaciones[[paste0(«c», i)]] <- df[, i]}
# Agregar combinaciones de tamaño 2 hasta k.
contador <- k
for (i in 2:k) {
combinaciones <- combn(k, i)
for (j in 1:ncol(combinaciones)) {
idx <- combinaciones[, j]
contador <- contador + 1
lista_combinaciones[[paste0(«c», contador)]] <- rowSums(df[, idx])}}
return(lista_combinaciones)}
lista_x <- generar_combinaciones_lineales(datos_orig)
# Cálculo de los parámetros comunes para la estandarización.
u <- log(log(n)) – log(n)
mu <- 1.0521 * u – 1.2725
v <- log(log(n)) + 2 / log(n)
sigma <- -0.26758 * v + 1.0308
cat(«\nParámetros comunes para la estandarización de las», 2^k – 1, «variables creadas por combinación lineal de las», k, «variables originales:\n»)
cat(«\nValor esperado de ln(1-W’): μ =», mu, «.\n»)
cat(«Desviación estándar de ln(1-W’): σ =», sigma, «.\n»)
# Aplicar prueba y guardar en tabla.
resultados <- lapply(names(lista_x), function(nombre) {
datos <- lista_x[[nombre]]
resultado <- shapiro.test(datos)
z <- (log(1 – resultado$statistic) – mu) / sigma
p <- 1 – pnorm(z)
Normalidad <- if (resultado$p.value < alpha) «No» else «Si»
return(c(nombre, round(resultado$statistic, 4), round(z, 4), round(p, 4), Normalidad))
})
# Convertir a marco de datos (data frame).
tabla_resultados <- as.data.frame(do.call(rbind, resultados), stringsAsFactors = FALSE)
colnames(tabla_resultados) <- c(«Variable», «W'», «z'», «p», «Normalidad»)
# Mostrar tabla.
cat(«\nTabla. Prueba de normalidad univariante de Shapiro y Wilk con la estandarización de Royston para las», 2^k-1, «combinaciones lineales de variables\n»)
print(tabla_resultados, row.names = FALSE)
cat(«\nNota. Variable = suma de la correspondiente combinación sin repetición de las variables; W’ = estadística W’ de Shapiro y Francia, z’ = valor estandarizado de W’ mediante la estandarización de Royston; p = valor de probabilidad a la cola derecha de una distribución normal estándar; y Normalidad univariante: Sí = p ≥ α =», alpha, «, se mantiene la hipótesis nula de normalidad, y No = p <«, alpha, «, se rechaza.\n»)
# Función para calcular q’.
calcular_q <- function(lista_datos) {
z_vals <- sapply(lista_datos, function(x) {
resultado_sf <- sf.test(x)
z <- (log(1 – resultado_sf$statistic) – mu) / sigma
return(as.numeric(z))
})
z_values <- ifelse(z_vals < 0, 0, z_vals)
a <- sum(z_vals < 0)
q <- sum(z_values^2)
return(list(q = q, a = a, z_vals = z_vals, z_values = z_values))
}
# Valor de probabilidad con la distribución chi-cuadrado estándar.
resultado_q <- calcular_q(lista_x)
q_stat <- resultado_q$q
a <- resultado_q$a
nc <- length(lista_x)
# gl_chisq <- nc – a # con corrección por valores truncados.
gl_chisq <- nc # sin corrección por valores truncados.
q_crit <- qchisq(1 – alpha, df = gl_chisq)
p_value <- pchisq(q_stat, df = gl_chisq, lower.tail = FALSE)
power <- 1 – pchisq(q_crit, df = gl_chisq, ncp = q_stat, lower.tail = TRUE)
# Resultados QSFa.
cat(«\nNúmero de combinaciones entre las», k, «variables originales: nc =», nc, «.\n»)
cat(«Número de valores z’ anulados por ser negativos: a =», a, «.\n»)
cat(«Grados de libertad: gl =», gl_chisq, «.\n»)
cat(«\nEstadística de contraste: q’ =», round(q_stat, 4), «.\n»)
cat(«\nValor de probabilidad y valor crítico desde una distribución chi-cuadrado estándar:\n»)
cat(«\nValor crítico o cuantil de orden», 1 – alpha,» de una distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad: 1-αχ²[gl] =», round(q_crit, 4), «.\n»)
if (q_stat <= q_crit) {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», alpha, «con base en el valor crítico desde la aproximación.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», alpha, «con base en el valor crítico desde la aproximación chi-cuadrado.\n»)}
cat(«Valor de probabilidad a la cola derecha en una distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad: p =», round(p_value, 4), «.\n»)
if (p_value < alpha) {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», alpha, «con base en el valor de probabilidad desde la aproximación.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», alpha, «con base en el valor de probabilidad desde la aproximación chi-cuadrado.\n»)}
cat(«Potencia estadística desde la aproximación chi-cuadrado para un nivel de significancia de», alpha, «: phi =», power, «.\n»)
cat(«Si se rechaza la hipótesis nula, la potencia estadística debería ser superior a 0.5, preferiblemente mayor que 0.8; mientras que, si no se rechaza la hipótesis nula, debería ser inferior a 0.5, preferiblemente menor que 0.2. En otro caso, el resultado es contradictorio o cuestionable.\n»)
# Contraste de la independencia serial en los valores z’.
z_w <- resultado_q$z_values
cat(«\nContraste de la independencia entre los», nc, «valores de W’ logarítmicamente transformados, estandarizados y truncados (a 0 cuando son negativos), usados para calcular q'(z_w).\n»)
RunsTest(z_w, y = NULL, alternative = «two.sided», exact = TRUE, correct = FALSE)
lag_max <- min(10, round(nc/5, 0)) # Regla de Hyndman y Athanasopoulos (2021) para determinar el rezago máximo en series temporales no estacionarias.
# lag_max <- 12 * (n/100)^0.25 # Regla de Schwert para determinar el rezago máximo.
ljung_box_test <- lapply(1: lag_max, function(k) Box.test(z_w, lag = k, type = «Ljung-Box», fitdf = 0))
ljung_box_tabla <- data.frame(
Rezago = 1:lag_max,
Chi_cuadrado = sapply(ljung_box_test, function(x) round(x$statistic, 4)),
gl = sapply(ljung_box_test, function(x) round(x$parameter, 4)),
p = sapply(ljung_box_test, function(x) round(x$p.value, 4))
)
cat(«\nTabla. Prueba de Ljung y Box de independencia serial en la secuencia de valores z_w_prima\n»)
print(ljung_box_tabla)
cat(«\nNota. Rezago = intervalo entre los valores que conforman los pares de datos en la autocorrelación, X² = estadística de contraste, gl = grados de libertad y p = probabilidad a la cola derecha en la distribución chi-cuadrado estándar.\n»)
# Función bootstrap preservando las correlaciones de la muestra original.
resample_preserving_corr <- function(df, B) {
replicate(B, {
df[sample(1:nrow(df), size = nrow(df), replace = TRUE), ]
}, simplify = FALSE)
}
# Función para generar lista de combinaciones.
generar_lista_boot <- function(df) {
lista_boot <- setNames(as.list(df), paste0(«c», 1:ncol(df)))
for (i in 2:ncol(df)) {
combinaciones <- combn(ncol(df), i)
for (j in 1:ncol(combinaciones)) {
idx <- combinaciones[, j]
nombre <- paste0(«c», length(lista_boot) + 1)
lista_boot[[nombre]] <- rowSums(df[, idx])
}
}
return(lista_boot)}
# Simulaciones bootstrap preservando correlaciones.
set.seed(123) # Se fija una semilla para reproducibilidad
B <- 1000 # Número de remuestreos con reposición desde la muestra original
boot_datasets <- resample_preserving_corr(datos_orig, B)
q_boot_vals <- sapply(boot_datasets, function(df_boot) {
lista_boot <- generar_lista_boot(df_boot)
calcular_q(lista_boot)$q
})
# Probabilidad desde la distribución bootstrap empírica.
boot_p_value <- mean(q_boot_vals >= q_stat)
cat(«\nDistribución bootstrap (con correlación conservada) empírica o desde la muestral original:\n»)
cat(«Número de muestras bootstrap: B =», B, «.\n»)
cat(«Valor de probabilidad bootstrap a la cola derecha en la distribución bootstrap empírica: p_boot =», round(boot_p_value, 4), «.\n»)
if (boot_p_value < alpha) {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», alpha, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap empírica.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», alpha, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap empírica.\n»)}
# Generar una muestra normativa con probabilidades equiespaciadas y la transformación Cholesky de la matriz de correlaciones de la muestra original.
p <- seq(0.5 / n, 1 – 0.5 / n, length.out = n)
datos_indep <- matrix(NA, nrow = n, ncol = k)
set.seed(123) # Se fija una semilla para reproducibilidad
for (j in 1:k) {datos_indep[, j] <- qnorm(sample(p))}
mat_cor <- cor(datos_orig)
mat_chol <- chol(mat_cor)
datos_normativos <- datos_indep %*% mat_chol
# Definir las variables-suma de todas las posibles combinaciones entre las variables originales.
lista_x_norm <- generar_combinaciones_lineales(datos_normativos)
result_q_norm <- calcular_q(lista_x_norm)
# Generar distribución bootstrap normativa con remuestreo de filas completas.
B <- 1000 # Número de remuestreos con reposición desde la muestra normativa
q_boot_norm_vals <- replicate(B, {
indices <- sample(1:n, size = n, replace = TRUE)
muestra_boot <- datos_normativos[indices, ]
lista_boot_n <- generar_combinaciones_lineales(muestra_boot)
calcular_q(lista_boot_n)$q
})
# Valor de probabilidad y valor crítico en la distribución bootstrap normativa.
boot_norm_p_value <- mean(q_boot_norm_vals >= q_stat)
# orden_cuantil <- 1-alpha # Sin compensación por cola derecha muy pesada.
orden_cuantil <- 1-2*alpha # Con compensación por cola derecha muy pesada.
valor_critico_boot_norm <- quantile(q_boot_norm_vals, probs = orden_cuantil)
cat(«\nDistribución bootstrap normativa (con correlación conservada):\n»)
cat(«Número de muestras bootstrap: B =», B, «.\n»)
cat(«Valor de la estadística q’ en la muestra normativa: q_mvn_boot =», result_q_norm$q, «.\n»)
cat(«Media de la distribución bootstrap normativa: m(mvn_boot_dist) =», mean(q_boot_norm_vals), «.\n»)
cat(«Mediana de la distribución bootstrap normativa: mdn(mvn_boot_dist) =», median(q_boot_norm_vals), «.\n»)
p_value_mdn_norm <- 2 * min(mean(q_boot_vals >= median(q_boot_norm_vals)), mean(q_boot_vals <= median(q_boot_norm_vals)))
cat(«Probabilidad de que la distribución bootstrap empírica esté centrada en la mediana de la distribución bootstrap normativa: p_2 colas =», round(p_value_mdn_norm, 4), «.\n»)
if (p_value_mdn_norm < alpha) {cat(«Se rechaza hipótesis nula de medianas equivalentes entre las distribuciones bootstrap normativa y empírica en un contraste a dos colas con un nivel de significancia de», 1-orden_cuantil, «.\n»)
} else {cat(«Se mantiene hipótesis nula de medianas equivalentes entre las distribuciones bootstrap normativa y empírica en un contraste a dos colas con un nivel de significancia de «, 1-orden_cuantil, «.\n»)}
cat(«Valor crítico bootstrap o cuantil de orden», orden_cuantil, «de la distribución bootstrap normativa: q’_crit =», round(valor_critico_boot_norm, 4), «.\n»)
if (q_stat <= valor_critico_boot_norm) {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», 1-orden_cuantil, «con base en el valor crítico bootstrap (normativo).\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», 1- orden_cuantil, «con base en el valor crítico bootstrap (normativo).\n»)}
cat(«Valor de probabilidad a la cola derecha en la distribución bootstrap normativa: p_mvn_boot =», round(boot_norm_p_value, 4), «.\n»)
if (boot_norm_p_value < 1-orden_cuantil) {cat(«La hipótesis nula de normalidad multivariante se rechaza a un nivel de significancia de», 1-orden_cuantil, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap normativa.\n»)
} else {cat(«La hipótesis nula de normalidad multivariante se mantiene a un nivel de significancia de», 1-orden_cuantil, «con base en el valor de probabilidad bootstrap calculado mediante la distribución bootstrap normativa.\n»)}
boot_power <- mean(q_boot_vals > valor_critico_boot_norm)
cat(«Potencia estadística bootstrap para un nivel de significancia de», 1-orden_cuantil, «: phi_boot =», boot_power, «.\n»)
# Representación de las distribuciones muestral bootstrap (empírica y normativa).
# Quitar el símbolo hashtag precedente para grabar como un archivo JPG o TIFF.
# jpeg(«Hist_curva_dens2.jpg», width = 1200, height = 900, units = «px», res = 300)
# tiff(«Hist_curva_dens2.tiff», width = 1200, height = 900, units = «px», res = 300)
# par(mar = c(4.5, 4.5, 0.5, 0.5), cex.axis = 0.8)
# Curva de densidad de la distribución chi-cuadrado estándar como gráfico base.
x_seq <- seq(0, max(qchisq(0.999, df = gl_chisq), max(q_boot_vals) + 1, q_stat +1, max(q_boot_norm_vals) + 1), length.out = 1000)
y_chi_sq <- dchisq(x_seq, df = gl_chisq)
plot(x_seq, y_chi_sq, type = «l», lwd = 3, col = «darkgreen»,
main = «», xlab = «Valores q_prima», ylab = «Densidad»,
xlim = c(0, max(qchisq(0.999, df = gl_chisq), max(q_boot_vals), q_stat +1, max(q_boot_norm_vals))),
ylim = c(0, max(density(q_boot_vals)$y, y_chi_sq)))
# Histograma amarillo de la distribución bootstrap empírica de la estadística de contraste q’.
hist(q_boot_vals, breaks = 20, freq = FALSE, col = rgb(1, 1, 0, 0.5), border = «yellow2», add = TRUE)
# Curva amarilla de densidad de la distribución bootstrap empírica de la estadística de contraste q’.
lines(density(q_boot_vals), col = «yellow3», lwd = 2)
# Curva roja de densidad bootstrap normativa.
lines(density(q_boot_norm_vals), col = «red», lwd = 2)
# Línea vertical violeta para el valor de la estadística de contraste q’ observado.
abline(v = q_stat, col = «purple», lwd = 2, lty = 2)
# Línea vertical roja para el valor crítico normativo bootstrap.
abline(v = valor_critico_boot_norm, col = «red», lwd = 2, lty = 2)
# Línea vertical verde para el valor crítico de la distribución chi-cuadrado estándar.
abline(v = qchisq(1-alpha, df = gl_chisq), col = «darkgreen», lwd = 2, lty = 2)
# dev.off() # Quitar el símbolo hashtag cuando se graba la figura.
cat(«\nFigura 2. Histograma (amarillo) con las curvas de densidad de las distribuciones bootstrap empírica (amarilla), bootstrap normativa (roja) y chi-cuadrado estándar (verde). El valor de la estadística de contraste q se representa mediante una línea vertical violeta (q’ =», q_stat, «), el valor crítico de la distribución bootstrap normativa mediante una línea vertical roja («, orden_cuantil, «_q_mvn_boot =», valor_critico_boot_norm, «) y el valor crítico de la distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad mediante una línea vertical verde («, 1-alpha, «χ²[«, gl_chisq, «] =», q_crit, «).\n»)
cat(«\nNota. Histograma amarillo (regla de la Universidad de Rice) = densidades de la distribución bootstrap empírica de la estadística de contraste q’,
curva amarilla = curva de densidad de la distribución bootstrap empírica de la estadística de contraste q’,
curva roja = curva de densidad de la distribución bootstrap normativa (generada desde variables normalmente distribuidas con la misma estructura correlacional que las originales) de la estadística de contraste q’,
curva verde = distribución chi-cuadrado estándar con», gl_chisq, «grados de libertad,
línea vertical amarilla = valor de la estadística de contraste q’ en la muestra original,
línea vertical roja = valor crítico o cuantil de orden», orden_cuantil, «de la distribución bootstrap normativa,
línea vertical verde = valor crítico o cuantil de orden», 1-alpha, «de una distribución chi-2 estándar con», gl_chisq, «grados de libertad.\n»)
# Prueba H de normalidad multivariante de Royston (1983) para muestras de tamaño de 10 a 2000 con k mediciones.
# Paquete requerido.
library(MVN)
# El paquete MVN requiere R ≥ 3.5.0
# Si no se tiene el paquete, seleccionar espejo más próximo: chooseCRANmirror()
# install.packages(«MVN»)
# Lista de variables originales.
x1 <- c(7, 6, 4, 5, 3, 9, 5, 3, 3, 5, 5, 6, 5, 9, 6, 10, 8, 7, 7, 3, 6, 5, 8, 7, 1, 4, 7, 2, 6, 4, 4, 8, 6, 2, 1, 4)
x2 <- c(6, 7, 4, 4, 5, 7, 6, 5, 3, 4, 7, 5, 1, 8, 5, 10, 8, 6, 8, 2, 4, 2, 7, 9, 3, 3, 5, 3, 5, 4, 6, 7, 6, 6, 1, 4)
x3 <- c(7, 9, 5, 5, 6, 9, 5, 5, 6, 2, 6, 7, 2, 10, 6, 9, 9, 3, 8, 2, 4, 4, 9, 6, 2, 3, 7, 4, 3, 6, 4, 7, 6, 5, 1, 4)
x4 <- c(8, 7, 6, 7, 3, 9, 4, 2, 3, 4, 5, 5, 1, 10, 6, 8, 9, 2, 6, 2, 6, 4, 7, 7, 5, 5, 7, 5, 5, 4, 6, 6, 8, 3, 1, 4)
x5 <- c(6, 7, 4, 6, 3, 6, 8, 2, 4, 4, 8, 8, 4, 10, 6, 8, 7, 4, 6, 2, 2, 4, 9, 9, 3, 4, 4, 6, 4, 6, 6, 7, 7, 3, 1, 3)
x6 <- c(8, 6, 4, 7, 5, 9, 8, 3, 5, 7, 6, 6, 5, 10, 4, 10, 9, 4, 6, 3, 6, 2, 8, 8, 5, 3, 7, 7, 4, 5, 6, 7, 5, 3, 2, 4)
datos_orig <- data.frame(x1, x2, x3, x4, x5, x6)
n <- length(x1)
k <- length(datos_orig)
alpha <- 0.05
result <- mvn(datos_orig, subset = NULL, mvnTest = «royston»)
# Potencia estadística para H.
R <- cor(datos_orig)
lambda <- 5
mu <- 0.715
ln_n <- log(n)
v_n <- 0.21364 + 0.015124 * ln_n^2 – 0.0018034 * ln_n^3
r_ij <- R[lower.tri(R)]
c_ij <- r_ij^lambda * (1 – (mu / v_n) * (1 – r_ij)^mu)
c_bar <- mean(c_ij)
df_H <- k / (1 + (k – 1) * c_bar)
power_H_5 <- 1 – pchisq(qchisq(1-alpha, df = df_H), df = df_H, ncp = result$multivariateNormality$H, lower.tail = TRUE)
power_H_10 <- 1 – pchisq(qchisq(1-2*alpha, df = df_H), df = df_H, ncp = result$multivariateNormality$H, lower.tail = TRUE)
# Mostrar resultados.
cat(«\nTamaño muestral: n =», n, «.\n»)
cat(«Número de variables en la muestra original: k =», k, «.\n»)
cat(«Media de las correlaciones entre las variables: m(R) =», round(mean(r_ij), 4), «.\n»)
cat(«\nPrueba de normalidad H de Royston (1983)\n»)
cat(«Valor de la estadística de contraste: H =», round(result$multivariateNormality$H, 4), «.\n»)
cat(«Grados de libertad: gl =», df_H, «.\n»)
cat(«Valor de probabilidad para la hipótesis nula de normalidade multivariante: p =», result$multivariateNormality$p, «.\n»)
cat(«\nPotencia para H para un nivel de significancia de», alpha, «: phi =», round(power_H_5, 4), «.\n»)
cat(«Potencia para H para un nivel de significancia de», 2*alpha, «: phi =», round(power_H_10, 4), «.\n»)
cat(«Si se rechaza la hipótesis nula, la potencia estadística debería ser superior a 0.5, preferiblemente mayor que 0.8; mientras que, si no se rechaza la hipótesis nula, debería ser inferior a 0.5, preferiblemente menor que 0.2. En otro caso, el resultado es contradictorio o cuestionable.\n»)
#————————————————————————————————–
# Potencia relativa entre las pruebas H (Royston, 1983) y Q, evaluada mediante la aproximación chi-cuadrado y el enfoque bootstrap.
#————————————————————————————————–
# Para ejecutar correctamente esta parte del script, es necesario correr previamente, y de forma secuencial, el script del Apéndice 1 (estadísticas W de Shapiro y Wilk, 1965, estandarizadas por Royston, 1992) o el del Apéndice 2 (estadísticas W’ de Shapiro y Francia, 1972, estandarizadas por Royston, 1993).
# Dichos scripts generan los valores de: power (potencia bajo la aproximación chi-cuadrado) y boot_power (potencia obtenida mediante bootstrap)
# Después de ello, debe ejecutarse el script del Apéndice 3, que calcula power_H para la prueba H.
# Alternativamente, es posible introducir manualmente los valores obtenidos en una ejecución previa e independiente de los Apéndices 1 o 2. Los valores en azul son los de la muestra analizada de 36 6-tuplas.
# power <- 0.07598624 # Potencia de la aproximación chi-cuadrado de QSW con α = 0.05.
# boot_power <- 0.027 # Potencia según el enfoque bootstrap de QSW con α = 0.1.
# power <- 0.06032782 # Potencia de la aproximación chi-cuadrado de QSF con α = 0.05.
# boot_power <- 0.018 # Potencia según el enfoque bootstrap de QSF con α = 0.1.
# Eliminar el símbolo de numeral o hashtag (#) antes de power y boot_power (de QSW o QSF) si se desea visualizar el resultado de potencia relativa de este ejemplo específico al ejecutar de forma independiente el script del tercer apéndice.
relative_power1 <- power_H_5 / power
cat(«\nPotencia relativa (H / Q_aprox_Chi2) con un nivel de significancia de», alpha, «: Rel_phi =», round(relative_power1, 4), «.\n»)
cat(«Si se rechaza la hipótesis nula de normalidad multivariante (p < α = 0.05) y los valores de potencia son mayores que 0.5, valores de potencia relativaH / Q_aprox_Chi2 > 1 indican mayor potencia de la prueba H de Royston y < 1 indican mayor potencia de la prueba Q desde la aproximación chi-cuadrado.\n»)
cat(«Si se mantiene la hipótesis nula de normalidad multivariante (p ≥ α = 0.05) y los valores de potencia son menores que 0.5, valores de potencia relativa H / Q_aprox_Chi2 > 1 sugieren un mejor rendimiento de la prueba Q desde la aproximación chi-cuadrado y < 1 mejor rendimiento de la prueba H.\n»)
relative_power2 <- power_H_10 / boot_power
cat(«\nPotencia relativa (H / Q_boot) con un nivel de significancia de», 2*alpha, «: Rel_phi =», round(relative_power2, 4), «.\n»)
cat(«Si se rechaza la hipótesis nula de normalidad multivariante (p < α = 0.1) y los valores de potencia son mayores que 0.5, valores de potencia relativa H / Q_boot > 1 indican mayor potencia de la prueba H de Royston y < 1 mayor potencia de la prueba Q desde el enfoque bootstrap.\n»)
cat(«Si se mantiene la hipótesis nula de normalidad multivariante (p ≥ α = 0.1) y los valores de potencia son menores que 0.5, valores de potencia relativa H / Q_boot > 1 sugieren un mejor rendimiento de la prueba Q desde el enfoque bootstrap y < 1 mejor rendimiento de la prueba H.\n»)
Los tres apéndices pueden consultarse en el siguiente repositorio de GitHub de acceso abierto: https://github.com/josemoraldelarubia579/Script-de-R-para-la-Prueba-Q-de-Normalidad-Multivariante.git
Nombre del repositorio: Script de R para la Prueba Q de Normalidad Multivariante